REVIEW
프로바이오틱 유산균 발효조건 탐색을 위한 다반응 최적화의 활용
임성수
1
,
*

Applying Multi-Response Optimization to Explore Fermentation Conditions of Probiotics
Sungsue Rheem
1
,
*

Author Information & Copyright ▼
1Division of Big Data Science, Korea University, Sejong, Korea
*Corresponding author : Sungsue Rheem, Division of Big Data Science, Korea University, Sejong, Korea, Tel : +82-44-860-1550 Fax : +82-44-860-1551 E-mail :
rheem@korea.ac.kr
© Copyright 2023, Korean Society of Dairy Science and Biotechnology. This is an Open Access article distributed under the terms of the
Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits
unrestricted non-commercial use, distribution, and reproduction in any
medium, provided the original work is properly cited.
Received: Feb 12, 2023; Revised: Feb 22, 2023; Accepted: Feb 23, 2023
Published Online: Jun 30, 2023
Abstract
This review serves two purposes: first, to promote the use of improved optimization techniques in response surface methodology (RSM); and second, to enhance the optimum conditions for the fermentation of probiotics. According to research in dairy science, Lactiplantibacillus plantarum K79 is a candidate probiotic that has beneficial health effects, such as lowering blood pressure. The optimum conditions for L. plantarum K79 to produce peptides with angiotensin-converting enzyme (ACE) inhibitory activity were proposed, through modeling each of ACE inhibitory activity and pH as a function of the four factors that are skim milk concentration (%), incubation temperature (°C), incubation time (hours), and starter added amount (%). To estimate optimum conditions, the researchers employed a desirability-based multi-response optimization approach, utilizing third-order models with a nonsignificant lack of fit. The estimated optimum fermentation conditions for L. plantarum K79 were as follows: a skim milk concentration of 10.76%, an incubation temperature of 36.9°C, an incubation time of 23.76 hours, and a starter added amount of 0.098%. Under these conditions, the predicted ACE inhibitory activity was 91.047%, and the predicted pH was 4.6. These predicted values achieved the objectives of the multi-response optimization in this study.
Keywords: response surface methodology; lack of fit; third-order model; desirability; multi-response optimization
서 론
반응표면 방법론(response surface methodology, RSM)은 실험의 설계, 반응의 모형화 및 요인수준 조합의 최적화를 위한 통계적 기법들의 모임[1]으로, 낙농식품응용생물학을 포함하는 실험과학 분야에서 널리 활용되어 왔다. RSM에서의 대표적인 실험설계는 중심 합성 설계(central composite design[2])이고, 대표적인 분석모형은 2차 다항 모형(second-order polynomial model)이다.
실제 실험연구 상황에서는 둘 이상의 반응들을 동시에 최적화하는 다반응 최적화가 요구되는 경우가 많은데, 가능한 한 정확하게 다반응 최적화를 실행하기 위해서는 반응들에 대해 적합한 모형화를 실시하는 것이 필요하다. 다반응 최적화를 위한 모형화는 각 반응에 대한 모형이 전보[3]에서 제시된 기준들 중에서 아래의 기준 (1), (2), (3)을 만족시키고 추가적으로 기준 (4)를 모두 충족시킬 때, 적합한 모형화라고 할 수 있다:
-
(1) 모형이 5% 수준에서 유의함(모형의 p≤0.05).
-
(2) 적합 결여(lack of fit)가 5% 수준에서 유의하지 않거나(적합 결여 p>0.05) 적합 결여가 유의하더라도, 적합 결여 통계량의 분모인 순수오차 평균제곱(pure error mean square)이 매우 작은 경우에는, 적합 결여 유의성의 원인이 동일 실험 조건에서의 반응치들의 값들이 아주 비슷한 현상이어서, 적합 결여의 유의성이 문제가 되지 않음[4].
-
(3) 수정 결정계수가 0.8 이상임(adjusted r2≧0.8)[1].
-
(4) 각 실험 조건에서 다수의 반응들이 산출될 때에는, 각 반응에 대해 동일한 모형을 사용함[5].
Lactiplantibacillus plantarum K79 균주는 프로바이오틱스로 활용 가능성이 높은 유산균으로 최근의 연구에서 ACE 저해활성이 있는 것으로 보고되었다[6]. 이 연구에서 Park et al.[6]은 탈지유, 스타터 첨가량, 배양온도 및 배양시간을 종속변수로 하여 ACE 억제활성을 극대화시키는 최적 배양조건을 정립하였다. Park et al.[6]은 2차 다항 모형을 이용하여 배양 최적화를 하였는데, 반응이 두 개이어서 다반응 최적화(multi-response optimization)가 실시되었다. 두 개의 반응들 중 첫 번째 반응의 최적화에서의 목적은 반응을 목표치(target) 또는 그 이상으로 최대화하는 것(반응치가 target에 도달하면 만족스러운데, 그보다 더 크면, 클수록 더 좋음)이었다. 이 경우에
로 ‘반응 최대화 시 최적화 예측치 만족도’를 정의할 때, 첫 번째 반응의 ‘반응 최대화 시 최적화 예측치 만족도’는 96.3%로 100%에는 미치지 못하였다. 두 번째 반응의 최적화 예측치는 최적화 목표치와 일치했다.
본 연구에서는 Park et al.의 연구[6]에서 얻어진 실험 데이터를 활용하여 보다 개선된 2차 다항 모형화 방법을 제시하고 그 방법에 따라 2개의 반응들 각각에 대해 모형화를 실시한 다음, 그 모형화 결과들을 이용하여 2개의 반응들에 대한 다반응 최적화를 실시하였다. 이 다반응 최적화에서의 목적을 Park et al.의 연구[6]에서의 목적과 동일하게 했는데, 첫 번째 반응의 ‘반응 최대화 시 최적화 예측치 만족도’는 101.2%로 100%를 돌파하였다. 두 번째 반응의 최적화 예측치는 최적화 목표치와 일치했다. 물론, 최적화 예측치와 그 예측치를 산출하는 요인수준 조합을 찾아낸 후에는, 그 요인수준 조합에서의 확인실험이 필요하다.
본 론
1. 분석 대상 데이터
Park et al.[6]은 심혈관 질환의 예방과 치료 모두에 사용할 수 있는 보다 안전하고 혁신적이며 저렴한, L. plantarum K79를 이용한 ACE 억제제의 활성력과 pH를, 반응표면법을 통하여, 모형화하고 최적화하는 연구를 실시하였다. 본 연구에서는 그 연구[6]에서의 데이터를 보다 발전된 반응표면법으로 분석하여 개선된 모형화와 향상된 최적화를 제시하고자 한다. L. plantarum K79에 의해 발효되는 우유의, ACE 억제제의 활성력과 pH를 최적화하는 조건을 찾기 위한 Park et al.[6]의 연구에서의 실험 설계의 코드화된 요인들(X1, X2, X3, X4)과 그 수준들, 그리고 실제 요인들(F1, F2, F3, F4)과 그 수준들이 Table 1에 제시되어 있다.
Table 1.
Experimental design in coded and actual factors and responses
| Run |
Design point |
X1 |
X2 |
X3 |
X4 |
F1 |
F2 |
F3 |
F4 |
Y1 |
Y2 |
| 1 |
1 |
–1 |
–1 |
–1 |
–1 |
8 |
34.5 |
12 |
0.065 |
64.5 |
5.94 |
| 2 |
2 |
1 |
–1 |
–1 |
–1 |
12 |
34.5 |
12 |
0.065 |
78.1 |
5.93 |
| 3 |
3 |
–1 |
1 |
–1 |
–1 |
8 |
39.5 |
12 |
0.065 |
73.5 |
5.4 |
| 4 |
4 |
1 |
1 |
–1 |
–1 |
12 |
39.5 |
12 |
0.065 |
83.4 |
5.46 |
| 5 |
5 |
–1 |
–1 |
1 |
–1 |
8 |
34.5 |
20 |
0.065 |
80.1 |
4.85 |
| 6 |
6 |
1 |
–1 |
1 |
–1 |
12 |
34.5 |
20 |
0.065 |
86.5 |
5.07 |
| 7 |
7 |
–1 |
1 |
1 |
–1 |
8 |
39.5 |
20 |
0.065 |
75.2 |
4.73 |
| 8 |
8 |
1 |
1 |
1 |
–1 |
12 |
39.5 |
20 |
0.065 |
87.7 |
4.86 |
| 9 |
9 |
–1 |
–1 |
–1 |
1 |
8 |
34.5 |
12 |
0.155 |
72.1 |
5.67 |
| 10 |
10 |
1 |
–1 |
–1 |
1 |
12 |
34.5 |
12 |
0.155 |
77.3 |
5.81 |
| 11 |
11 |
–1 |
1 |
–1 |
1 |
8 |
39.5 |
12 |
0.155 |
68.5 |
5.25 |
| 12 |
12 |
1 |
1 |
–1 |
1 |
12 |
39.5 |
12 |
0.155 |
81.5 |
5.41 |
| 13 |
13 |
–1 |
–1 |
1 |
1 |
8 |
34.5 |
20 |
0.155 |
79.6 |
4.48 |
| 14 |
14 |
1 |
–1 |
1 |
1 |
12 |
34.5 |
20 |
0.155 |
89.4 |
4.63 |
| 15 |
15 |
–1 |
1 |
1 |
1 |
8 |
39.5 |
20 |
0.155 |
79.1 |
4.71 |
| 16 |
16 |
1 |
1 |
1 |
1 |
12 |
39.5 |
20 |
0.155 |
85.6 |
4.86 |
| 17 |
17 |
–2 |
0 |
0 |
0 |
6 |
37 |
16 |
0.11 |
78.4 |
4.84 |
| 18 |
18 |
2 |
0 |
0 |
0 |
14 |
37 |
16 |
0.11 |
82.9 |
5.08 |
| 19 |
19 |
0 |
–2 |
0 |
0 |
10 |
32 |
16 |
0.11 |
86.1 |
4.43 |
| 20 |
20 |
0 |
2 |
0 |
0 |
10 |
42 |
16 |
0.11 |
73.6 |
5.04 |
| 21 |
21 |
0 |
0 |
–2 |
0 |
10 |
37 |
8 |
0.11 |
39.7 |
6.39 |
| 22 |
22 |
0 |
0 |
2 |
0 |
10 |
37 |
24 |
0.11 |
89.5 |
4.4 |
| 23 |
23 |
0 |
0 |
0 |
–2 |
10 |
37 |
16 |
0.02 |
85.2 |
5.05 |
| 24 |
24 |
0 |
0 |
0 |
2 |
10 |
37 |
16 |
0.2 |
87.4 |
4.97 |
| 25 |
25 |
0 |
0 |
0 |
0 |
10 |
37 |
16 |
0.11 |
84.1 |
5.15 |
| 26 |
25 |
0 |
0 |
0 |
0 |
10 |
37 |
16 |
0.11 |
85.6 |
5.14 |
| 27 |
25 |
0 |
0 |
0 |
0 |
10 |
37 |
16 |
0.11 |
87.3 |
5.15 |
Download Excel Table
이 실험에서의 실제 요인들은 포도당 1%가 첨가된 skim milk 농도(F1), 배양온도(F2), 배양시간(F3), starter 첨가량(F4)이고, 5개로 이루어지는 각 요인수준은 –2, –1, 0, 1, 2로 코드화되었다. 이 실험의 설계는 Box & Wilson의 중심 합성 설계[2]로, Table 1에 주어져 있다.
이 실험에서의 반응변수들은 ACE 억제율(Y1)과 pH(Y2)로, Table 1에 주어져 있다. 이 ACE 억제율은 실험의 각 run에서의 발효원액에서 100배 희석하여 나타낸 값이다[6].
2. 각 반응의 모형화
데이터는 SAS 소프트웨어에 의해 분석되었다. 반응의 모형화를 위해서는 SAS/STAT[8]이 사용되었고, 다반응 최적화를 위해서는 SAS data-step 프로그래밍이 이용되었으며, 3D 표면도 작성을 위해서는 SAS/GRAPH[9]가 사용되었다.
1) ACE(angiotensin converting enzyme) 억제율(Y1)에 대한 2차 모형에 의한 모형화
요인이 4개이므로 1차항 4개, 교차항 6개, 제곱항 4개로 이루어지는 2차 모형을 먼저 ACE 억제율(Y1) 데이터에 적합시켜 본다. 이 2차 모형에 대한 분산분석 결과가 Table 2에 주어져 있다.
Table 2.
Analysis of variance for the second-order model on Y1
| Source |
DF |
Sum of squares |
Mean square |
F-value |
Pr>F-value |
| Model |
14 |
2,152.7838 |
153.7703 |
3.24 |
0.0240 |
| Error |
12 |
568.9725 |
47.4144 |
|
|
| Lack of fit |
10 |
563.8458 |
56.3846 |
22 |
0.0442 |
| Pure error |
2 |
5.1267 |
2.5633 |
|
|
| Corrected total |
26 |
2,721.7563 |
|
|
|
| Root MSE=6.8858, Dependent mean=79.3296, r2=0.7910, Adj r2=0.5471 |
Download Excel Table
Table 2는 Y1에 대한 이 2차 모형에 대하여 다음을 알려주고 있다.
-
(1) 모형이 5% 수준에서 유의함(모형의 p=0.0240≤0.05): 기준 충족
-
(2) 적합 결여가 5% 수준에서 유의함(적합 결여 p=0.0442<0.05): 기준 미충족
-
(3) 수정 결정계수가 0.8 미만임(adjusted r2=0.5471<0.8): 기준 미충족
Y1에 대한 이 2차 모형은 기준 (2), (3)을 충족시키지 못하므로, 적합한 모형이 아닌 것으로 판정된다.
2) pH(Y2)에 대한 2차 모형에 의한 모형화
요인이 4개이므로 1차항 4개, 교차항 6개, 제곱항 4개로 이루어지는 2차 모형을 먼저 pH(Y2) 데이터에 적합시켜 본다. 이 2차 모형에 대한 분산분석 결과가 Table 3에 주어져 있다.
Table 3.
Analysis of variance for the second-order model on Y2
| Source |
DF |
Sum of squares |
Mean square |
F-value |
Pr>F-value |
| Model |
14 |
5.70276 |
0.40734 |
8.21 |
0.0004 |
| Error |
12 |
0.59540 |
0.04962 |
|
|
| Lack of fit |
10 |
0.59533 |
0.05953 |
1,786.00 |
0.0006 |
| Pure error |
2 |
0.00007 |
0.00003 |
|
|
| Corrected total |
26 |
6.29816 |
|
|
|
| Root MSE=0.22275, Dependent mean=5.13704, r2=0.9055, Adj r2=0.7952 |
Download Excel Table
Table 3은 Y2에 대한 이 2차 모형에 대하여 다음을 알려주고 있다.
-
(1) 모형이 5% 수준에서 유의함(모형의 p=0.0004<0.05): 기준 충족
-
(2) 적합 결여가 5% 수준에서 유의함(적합 결여 p=0.0006<0.05): 기준 미충족으로 보이지만, 이 경우는 적합 결여 통계량의 분모인 순수오차 평균제곱(pure error mean square)이 0.00003으로 매우 작아, 적합 결여 유의성의 원인이 동일 실험 조건에서의 반응치들의 값들이 아주 비슷한 현상(Run 25, 26, 27에서 Y2=5.15, 5.14¸ 5.15)으로, 적합 결여의 유의성이 문제가 되지 않음. 따라서, 기준 충족.
-
(3) 수정 결정계수가 0.8 미만임(adjusted r2=0.7952<0.8): 기준 미충족
Y2에 대한 이 2차 모형은 기준 (3)을 충족시키지 못하므로, 적합한 모형이 아닌 것으로 판정된다.
Y1과 Y2에 대한 2차 모형들이 모두 적합한 모형이 아닌 것으로 판정되었으므로, 다음 단계로, 2차 모형에 세제곱항들을 추가한 3차 모형[10]을 Y1과 Y2 데이터에 대해 적합시킨다.
3) ACE(angiotensin converting enzyme) 억제율(Y1)에 대한 3차 모형에 의한 모형화
요인이 4개이므로 1차항 4개, 교차항 6개, 제곱항 4개, 세제곱항 4개로 이루어지는 3차 모형을 ACE 억제율(Y1) 데이터에 적합시켜 본다. 이 3차 모형에 대한 분산분석 결과가 Table 4에 주어져 있다.
Table 4.
Analysis of variance for a third-order model on Y1
| Source |
DF |
Sum of squares |
Mean square |
F-value |
Pr>F-value |
| Model |
18 |
2,672.0946 |
148.4497 |
23.91 |
<0.0001 |
| Error |
8 |
49.6617 |
6.2077 |
|
|
| Lack of fit |
6 |
44.5350 |
7.4225 |
2.90 |
0.2788 |
| Pure error |
2 |
5.1267 |
2.5633 |
|
|
| Corrected total |
26 |
2,721.7563 |
|
|
|
| Root MSE=2.49153, Dependent mean=79.32963, r2=0.9818, Adj r2=0.9407 |
Download Excel Table
Table 4는 Y1에 대한 이 3차 모형에 대하여 다음을 알려주고 있다.
-
(1) 모형이 5% 수준에서 유의함(모형의 p=0.0001 미만<0.05): 기준 충족.
-
(2) 적합 결여가 5% 수준에서 유의하지 않음(적합 결여 p=0.2788>0.05): 기준 충족.
-
(3) 수정 결정계수가 0.8 이상임(adjusted r2=0.9407>0.8): 기준 충족.
Y1에 대한 이 3차 모형은, 기준 (1)–(3)을 충족시키고 r2도 0.9818로 매우 높아서, 적합한 모형인 것으로 판정된다. 이 모형의 계수 추정치들(coefficient estimates; b0, b1, …, b444)과 관련 통계치들은 Table 5에 주어져 있다.
Table 5.
Coefficient estimates and related statistics in the third-order model on Y1
| Model term |
Coefficient estimate (b0, b1, …, b444) |
SE |
t-value |
p-value |
| Intercept |
85.66667 |
1.43848 |
59.55 |
<0.0001 |
| X1 |
6.03333 |
0.88089 |
6.85 |
0.0001 |
| X2 |
1.61667 |
0.88089 |
1.84 |
0.1038 |
| X3 |
1.20833 |
0.88089 |
1.37 |
0.2074 |
| X4 |
0.15833 |
0.88089 |
0.18 |
0.8618 |
| X12 |
–1.08229 |
0.53943 |
–2.01 |
0.0797 |
| X22 |
–1.28229 |
0.53943 |
–2.38 |
0.0447 |
| X32 |
–5.09479 |
0.53943 |
–9.44 |
<0.0001 |
| X42 |
0.33021 |
0.53943 |
0.61 |
0.5574 |
| X1X2 |
0.43125 |
0.62288 |
0.69 |
0.5083 |
| X1X3 |
–0.40625 |
0.62288 |
–0.65 |
0.5326 |
| X1X4 |
–0.49375 |
0.62288 |
–0.79 |
0.4508 |
| X1X3 |
–1.43125 |
0.62288 |
–2.30 |
0.0506 |
| X2X4 |
–0.89375 |
0.62288 |
–1.43 |
0.1892 |
| X3X4 |
0.26875 |
0.62288 |
0.43 |
0.6775 |
| X13 |
–1.22708 |
0.35962 |
–3.41 |
0.0092 |
| X23 |
–1.18542 |
0.35962 |
–3.30 |
0.0109 |
| X33 |
2.81042 |
0.35962 |
7.81 |
<0.0001 |
| X43 |
0.09792 |
0.35962 |
0.27 |
0.7923 |
Download Excel Table
4) pH(Y2)에 대한 3차 모형에 의한 모형화
요인이 4개이므로 1차항 4개, 교차항 6개, 제곱항 4개, 세제곱항 4개로 이루어지는 3차 모형을 ACE 억제율(Y2) 데이터에 적합시켜 본다. 이 3차 모형에 대한 분산분석 결과가 Table 6에 주어져 있다.
Table 6.
Analysis of variance for a third-order model on Y2
| Source |
DF |
Sum of squares |
Mean square |
F-value |
Pr>F-value |
| Model |
18 |
6.11921 |
0.33996 |
15.20 |
0.0003 |
| Error |
8 |
0.17895 |
0.02237 |
|
|
| Lack of fit |
6 |
0.17888 |
0.02981 |
894.42 |
0.0011 |
| Pure error |
2 |
0.00007 |
0.00003 |
|
|
| Corrected total |
26 |
6.29816 |
|
|
|
| Root MSE=0.14956, Dependent mean=5.13704, r2=0.9716, Adj r2=0.9077 |
Download Excel Table
Table 6은 Y2에 대한 이 3차 모형에 대하여 다음을 알려주고 있다.
-
(1) 모형이 5% 수준에서 유의함(모형의 p=0.0003<0.05): 기준 충족.
-
(2) 적합 결여가 5% 수준에서 유의함(적합 결여 p=0.0011<0.05): 기준 미충족으로 보이지만, 이 경우는 적합 결여 통계량의 분모인 순수오차 평균제곱(pure error mean square)이 0.00003으로 매우 작아, 적합 결여 유의성의 원인이 동일 실험 조건에서의 반응치들의 값들이 아주 비슷한 현상(Run 25, 26, 27에서 Y2=5.15, 5.14¸ 5.15)으로, 적합 결여의 유의성이 문제가 되지 않음. 따라서, 기준 충족.
-
(3) 수정 결정계수가 0.8 이상임(adjusted r2=0.9077>0.8): 기준 충족.
Y2에 대한 이 3차 모형은, 기준 (1)–(3)을 충족시키고 r2도 0.9716으로 상당히 높아서, 적합한 모형인 것으로 판정된다. 이 모형의 계수 추정치들(coefficient estimates; c0, c1, …, c444)과 관련 통계치들은 Table 7에 주어져 있다.
Table 7.
Coefficient estimates and related statistics in the third-order model on Y2
| Model term |
Coefficient estimate (c0, c1, …, c444) |
SE |
t-value |
p-value |
| Intercept |
5.14667 |
0.08635 |
59.6 |
<0.0001 |
| X1 |
0.06333 |
0.05288 |
1.20 |
0.2653 |
| X2 |
–0.19250 |
0.05288 |
–3.64 |
0.0066 |
| X3 |
–0.39083 |
0.05288 |
–7.39 |
<0.0001 |
| X4 |
–0.11167 |
0.05288 |
–2.11 |
0.0677 |
| X12 |
–0.01896 |
0.03238 |
–0.59 |
0.5744 |
| X22 |
–0.07521 |
0.03238 |
–2.32 |
0.0487 |
| X32 |
0.08979 |
0.03238 |
2.77 |
0.0242 |
| X42 |
–0.00646 |
0.03238 |
–0.20 |
0.8469 |
| X1X2 |
0.00000 |
0.03739 |
0.00 |
1.0000 |
| X1X3 |
0.01875 |
0.03739 |
0.50 |
0.6296 |
| X1X4 |
0.01250 |
0.03739 |
0.33 |
0.7467 |
| X1X3 |
0.12250 |
0.03739 |
3.28 |
0.0113 |
| X2X4 |
0.06125 |
0.03739 |
1.64 |
0.1400 |
| X3X4 |
–0.01500 |
0.03739 |
–0.40 |
0.6988 |
| X13 |
–0.00083 |
0.02159 |
–0.04 |
0.9702 |
| X23 |
0.08625 |
0.02159 |
4.00 |
0.0040 |
| X33 |
–0.02667 |
0.02159 |
–1.24 |
0.2518 |
| X43 |
0.02292 |
0.02159 |
1.06 |
0.3194 |
Download Excel Table
3. 바람직성 함수 기법에 의한 다반응 최적화
Park et al.[6]의 연구에서는 Y1의 최대화 target 값을 90으로 하고(Y1의 값이 90에 도달하면 만족스러운데, 90보다 더 크면, 클수록 더 좋음) Y2의 특정치 target 값을 4.6으로 하는 다반응 최적화가 2차 모형에 근거하여 실시되었는데, 그 최적화가 찾아낸 요인수준 조합에서의 Y1의 예측치는 86.6916으로, ‘반응 최대화 시 최적화 예측치 만족도’는 86.69/90=96.3%였고, Y2의 예측치는 4.6으로 특정치 target 값과 일치하였다.
우리의 연구에서는 Park et al.[6]의 연구에서와 같은 목적의 다반응 최적화가 3차 모형에 근거하여 실시되었는데, 그 결과는 Table 8에 나타나 있다. 이 다반응 최적화는 바람직성(desirability) 함수 기법[11]에 의해 실시되었는데, 이 기법은, 실험 영역(여기서는 X12 + X22 + X32 + X42 ≦ (±1)2 + (±1)2 + (±1)2 + (±1)2 = 4인 영역) 내의 모든 가능한 요인수준 조합들에서, 각 반응의 모형으로부터의 예측치에 개별 바람직성(individual desirability) 값을 반응값이 만족스러울수록 1에 가깝게 되도록(아주 만족스러우면 개별 바람직성 값이 1이 됨), 그리고 반응값이 불만족스러울수록 0에 가깝게 되도록(아주 불만족스러우면 개별 바람직성 값이 0이 됨) 정의하고, 전체 바람직성(overall desirability) 값을 개별 바람직성 값들의 기하평균으로 정의하여, 전체 바람직성 값을 최대화하는 요인수준 조합을 찾는 방법이다. 이를 위하여 grid 상 탐색이 이용되었다[7]. Table 8에서의 최적화 결과를 나타내는 수치들은 SAS data-step 프로그래밍에 의해 계산되었다. 전체 바람직성 값이 가질 수 있는 가장 큰 값은 1인데, Table 8에서의 최적화 결과에서의 전체 바람직성 값은 1이었다.
Table 8.
Multi-response optimization results
| X1 |
X2 |
X3 |
X4 |
F1= Skim milk conc. (%) |
F2= Incubation temp. (℃) |
F3= Incubation time (h) |
F4= Starter added amount (%) |
Predicted Y1 (=ACE inhibitory activity [%]) |
Predicted Y2 (=pH) |
| 0.38 |
–0.04 |
1.94 |
–0.26 |
10.76 |
36.9 |
23.76 |
0.0983 |
91.0468 |
4.6000 |
Download Excel Table
Table 8에 나타나 있는 바와 같이, 우리의 최적화가 찾아낸 최적 요인수준 조합에서의 Y1의 예측치는 91.0468로, ‘반응 최대화 시 최적화 예측치 만족도’는 91.0468/90=101.2%였고, Y2의 예측치는 4.6으로 특정치 target 값과 일치하였다. 즉, Y1과 Y2의 최적화 목표들이, 2차 모형에 근거한 다반응 최적화에서는 예측치 상으로 모두 달성되지는 못하였는데, 3차 모형에 근거한 다반응 최적화에서는 예측치 상으로 모두 달성된 것으로 계산되었고, 그 구체적인 내용으로, 3차 모형에 근거한 최적화에서의 Y1 예측치의 만족도(101.2%)가 2차 모형에 근거한 최적화에서의 Y1 예측치의 만족도(96.3%) 보다 더 우수한 것으로 나타났다. 물론, 최적화 예측치와 그 예측치를 산출하는 요인수준 조합을 찾아낸 후에는, 그 요인수준 조합에서의 확인실험이 필요하다.
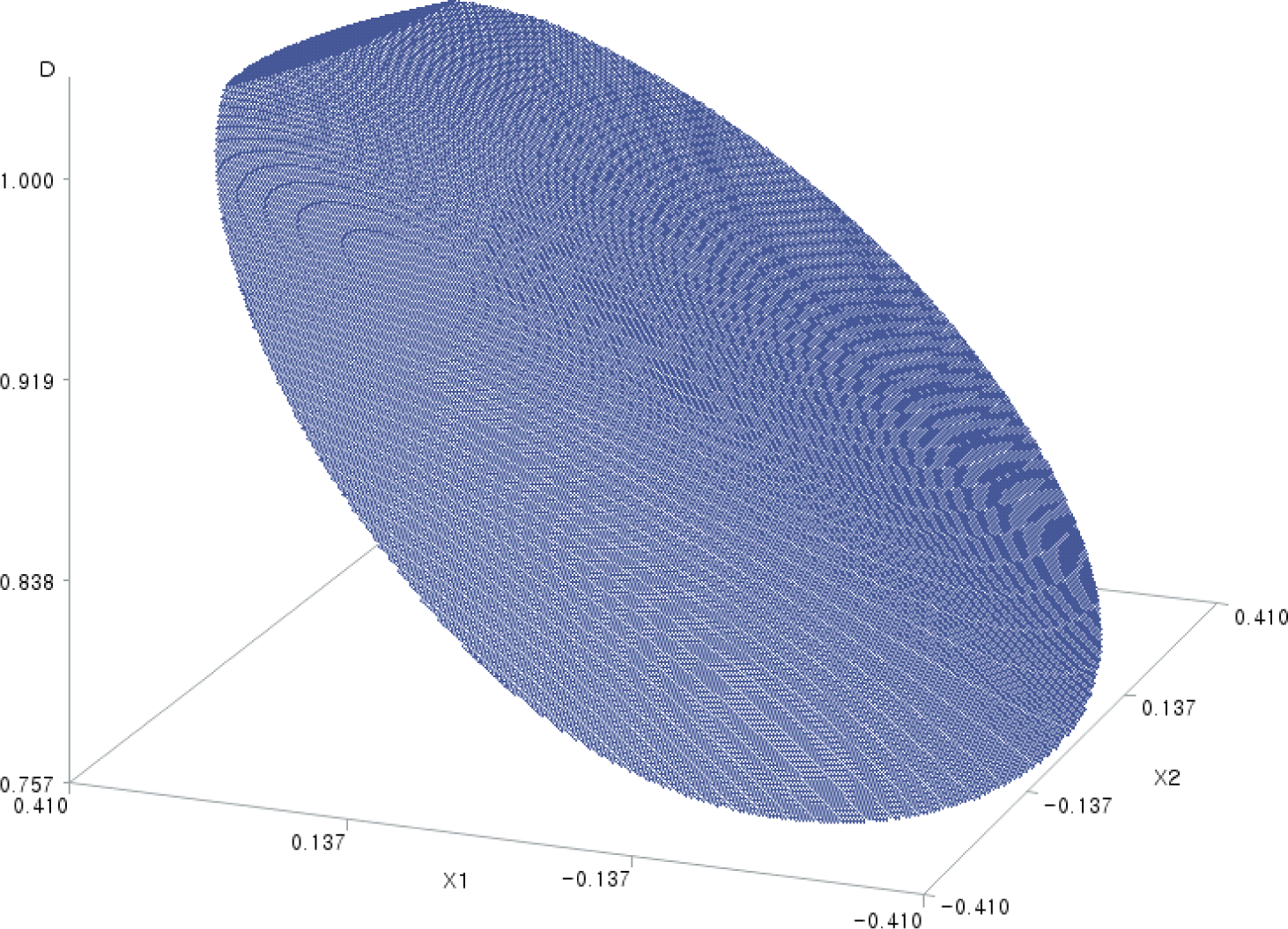
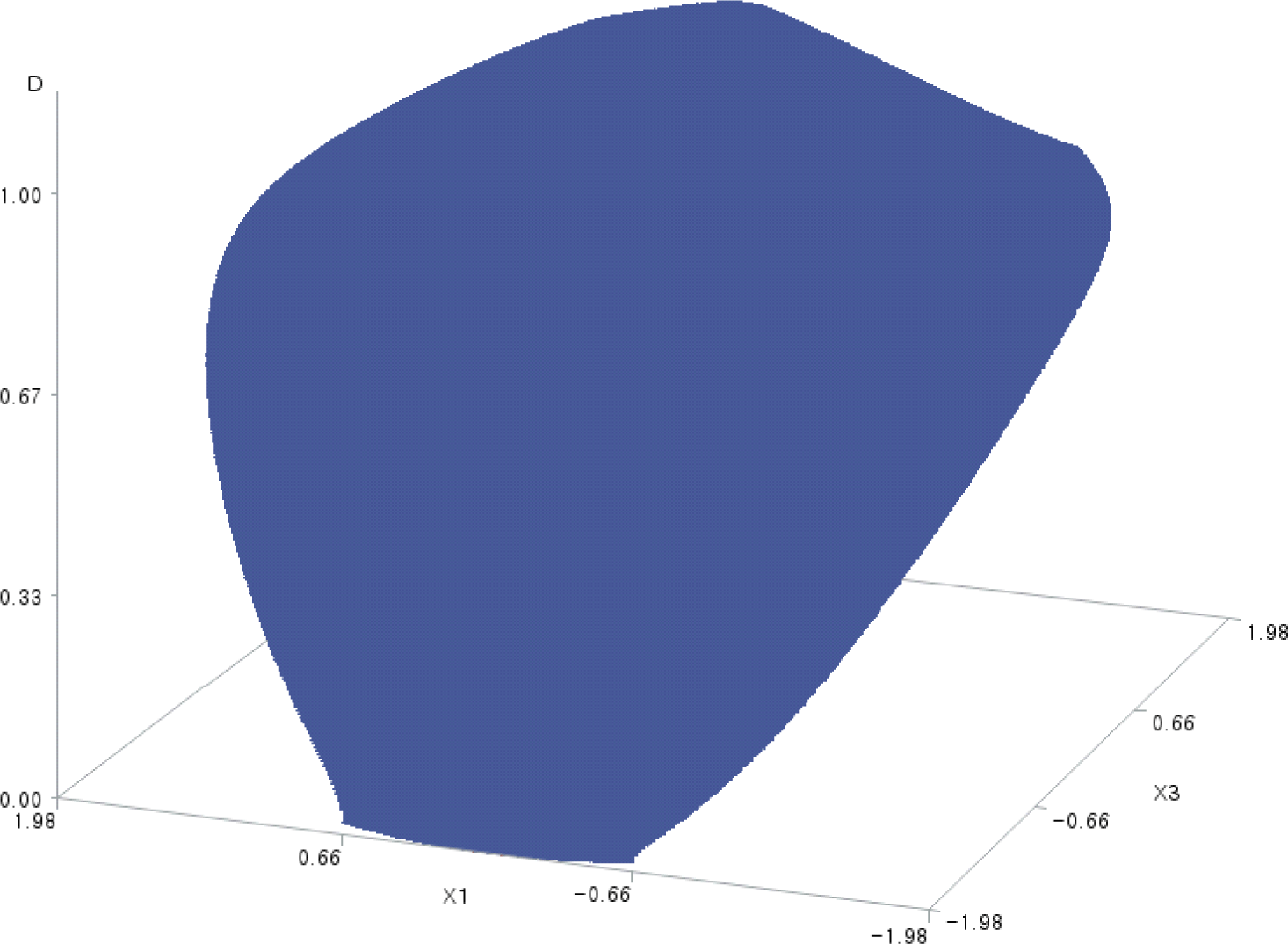
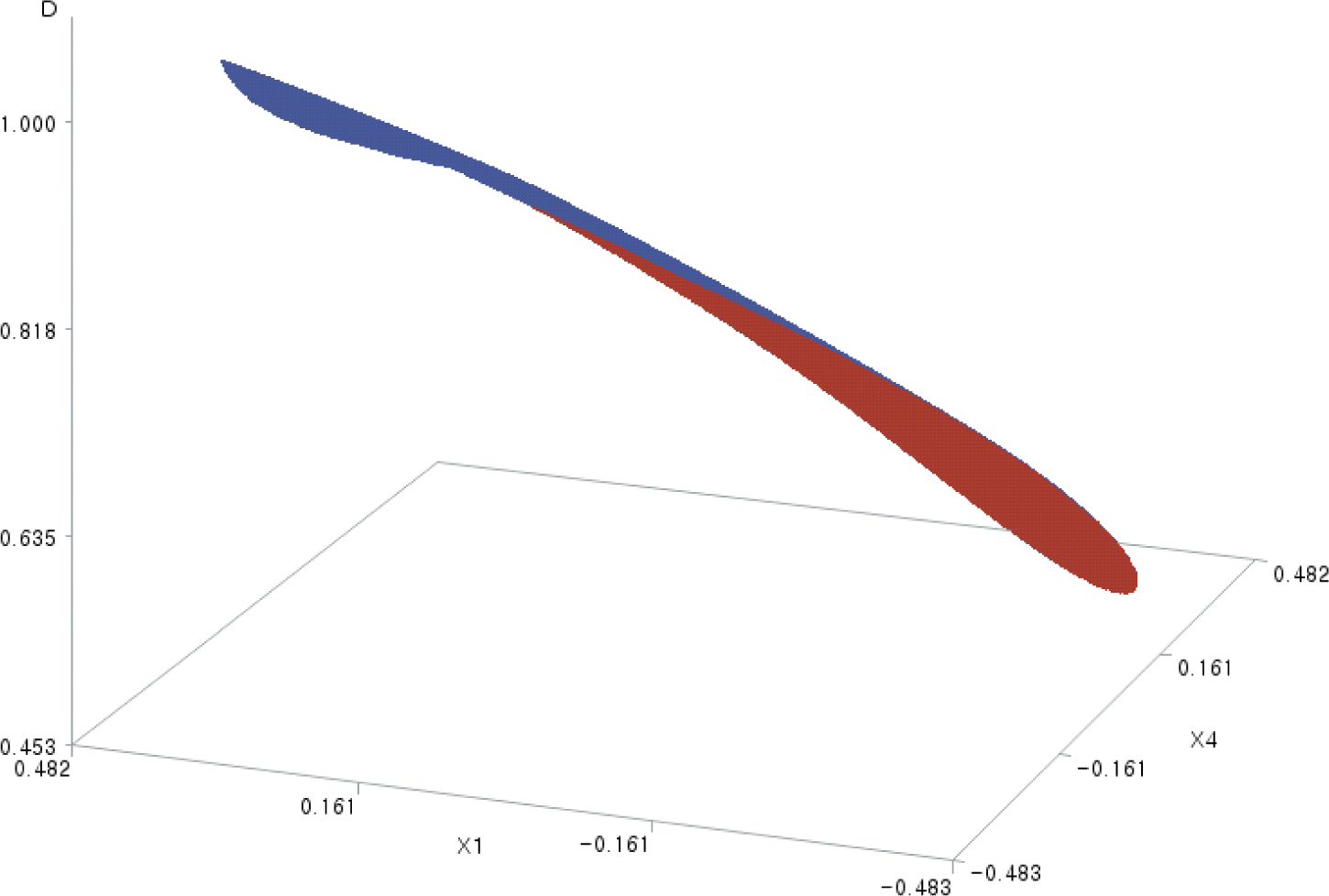
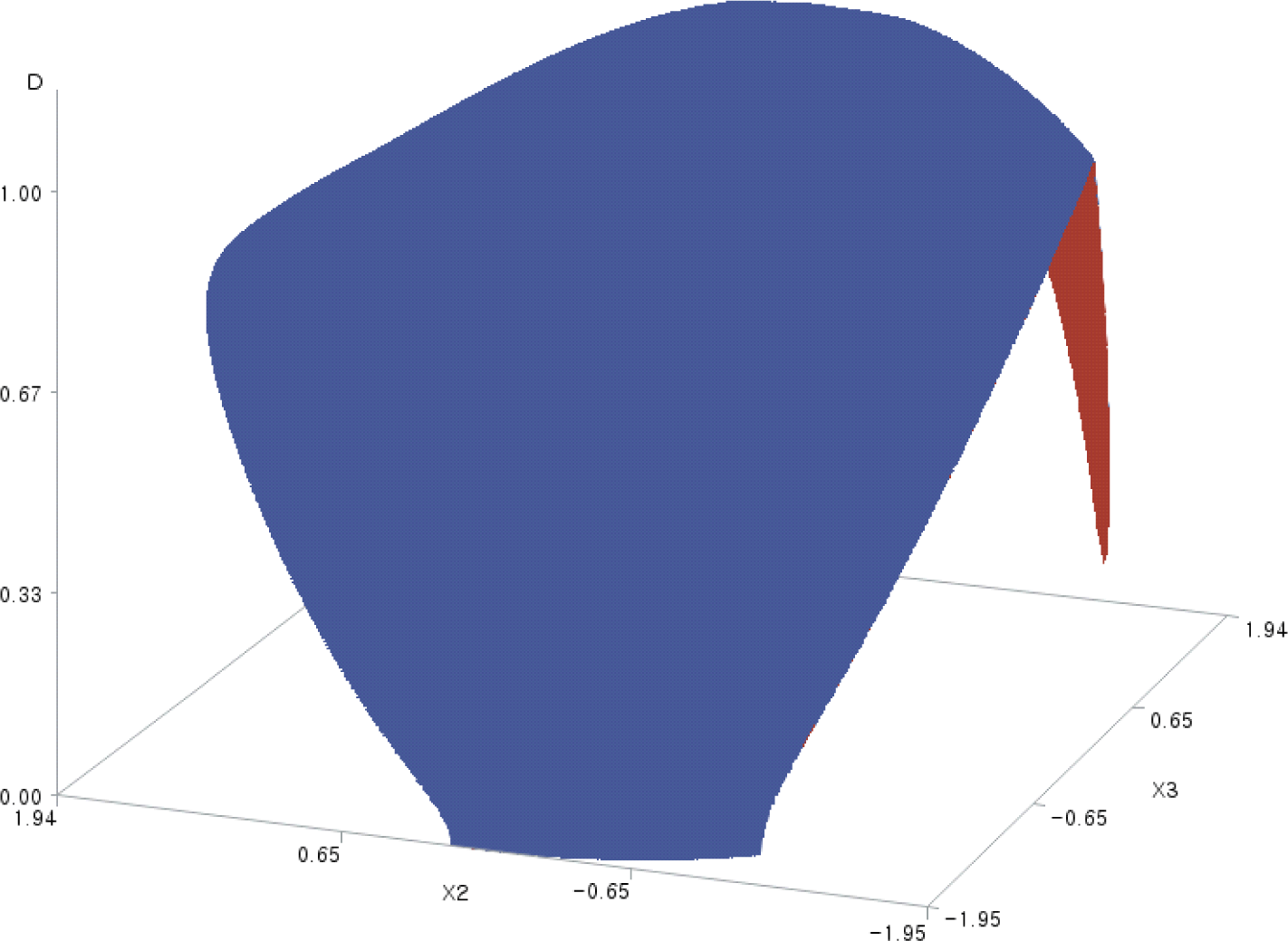
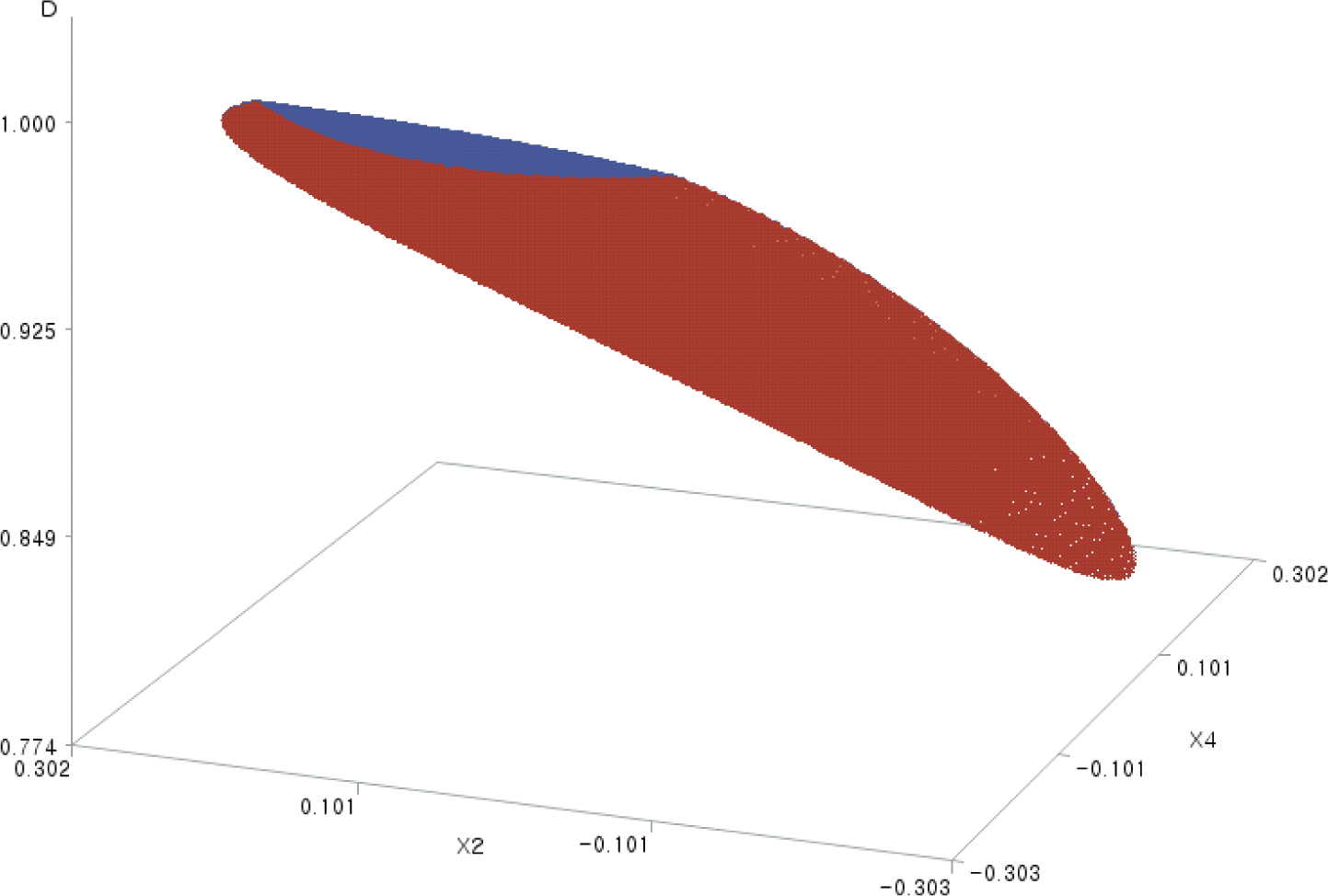
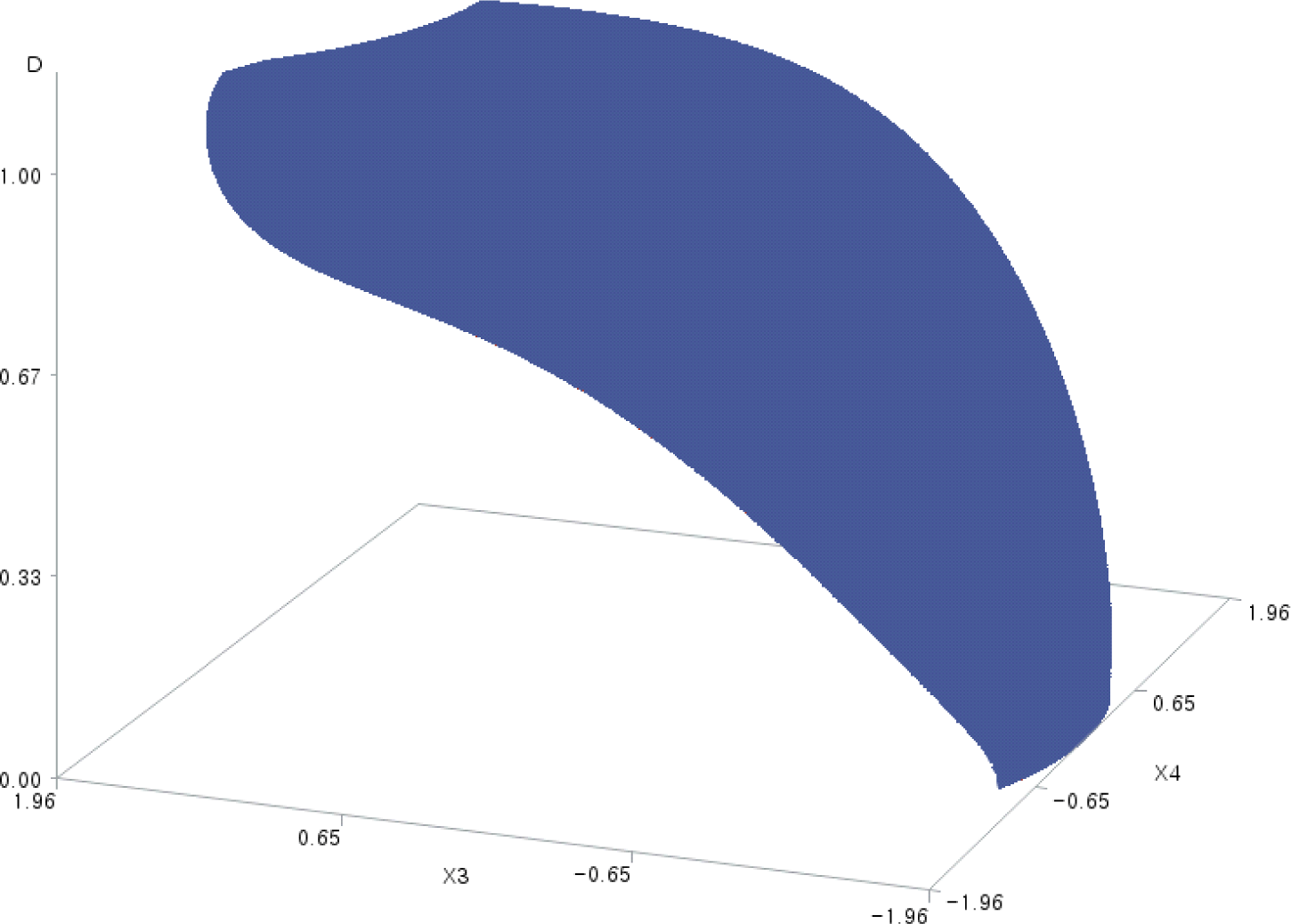
4. 3차원 표면도에 의한 전체 바람직성 함수의 시각화
코드화된 요인 2개씩 짝을 지어 각 가로축에 배치하고 전체 바람직성 예측치를 세로축에 배치한 3차원 표면도(3-dimensional surface plots) 6개를 SAS/GRAPH로 작성하여 각각 Figs. 1–6에 담았다. 이 그림들은 실험 영역(여기서는 X12 + X22 + X32 + X42 ≦ (±1)2 + (±1)2 + (±1)2 + (±1)2 = 4인 영역) 내에서 그려졌고, 그림에 표시되지 않은 X 변수들의 값은 Table 8에서의 값이다.
결 론
본 연구에서는 Park et al.[6]의 연구에서 시도된 프로바이오틱 유산균 발효조건에 대한 탐색이 반응표면방법론에서의 고급 기법을 활용하여 실시되었는데, ACE inhibitory activity(%)를 90 이상으로 최대한 높이고 pH를 4.6으로 맞추는 요인수준 조합으로 skim milk conc. 10.76%, incubation temperature 36.9℃, incubation time 23.76시간, starter added amount 0.0983%가 제시되었다. 이 요인수준 조합에서의 반응들의 예측치들은 ACE inhibitory activity 91.0468%, pH 4.6으로 계산되었고, 이 예측치들은 ACE inhibitory activity 90% 이상, pH 4.6이라는 다반응 최적화의 목표를 달성하는 값들이다. 물론 이 요인수준 조합은, 실제에서 활용되려면, 확인실험을 거쳐야 할 것이다.
본 연구는 또한 반응표면방법론에서의 고급 기법으로 2차 다항 모형의 적합 결여가 유의할 때의 분석 모형으로서 3차 모형을 활용하는 방법을 소개하였고, 바람직성 함수에 기반을 둔 다반응 최적화 기법도 설명하였으며, 특히, graphical display로서, 최적화 도구인 바람직성 함수의 3D 표면도도 제시하였다. 바람직성 함수의 3D 표면도는 직접 그리기가 쉽지 않아 보통 문헌에 잘 나타나지 않는 그림인데, 다반응 최적화에서의 각 요인의 역할을 이해하는 데에 도움을 준다.
2차 모형을 사용하는 단반응 또는 다반응의 최적화는 통계 소프트웨어 Minitab에서 ‘통계분석>실험계획법>반응 표면>반응 최적화 도구’ 절차를 이용하면 용이하게 실행할 수 있다.
본 연구를 통해서, 적합성이 개선된 모형에 기반한 최적화를 실시하면, 예측치 상으로 바람직성이 향상된 최적화 결과가 도출됨을 관찰하였다.
감사의 글
본 논문은 2022년도 고려대학교 공공정책대학 교내지원연구비에 의해 연구되었음.
References
Myers RH, Montgomery DC, Anderson-Cook CM. Response surface methodology: process and product optimization using designed experiments. 3rd ed. Hoboken, NJ: John Wiley & Sons; 2009.

Box GEP, Wilson KB. On the experimental attainment of optimum conditions. J R Stat Soc Series B Methodol. 1951;13:1-45.


Rheem S, Oh S. Applying response surface methodology to predict the homogenization efficiency of milk. J Dairy Sci Biotechnol. 2023;41:1-8.


Rheem S, Rheem I. The model-fit proportion and the coefficient of pure error variation, the measures to supplement the lack-of-fit test in response surface analysis. J Korean Data Anal Soc. 2012;14:2989-2994.

Rheem S. A suggestion on multiresponse modeling in response surface analysis. J Korean Data Anal Soc. 2006;8:2317-2328.

Park YK, Hong SP, Lim SD. Optimization of skim milk fermentation conditions by response surface methodology to improve ACE inhibitory activity using Lactiplantibacillus plantarum K79. J Dairy Sci Biotechnol. 2022;40:93-102.


SAS. SAS/STAT user’s guide. Release 9.4. Cary, NC: SAS Institute; 2016.

SAS. SAS/GRAPH user’s guide. Release 9.4. Cary, NC: SAS Institute; 2016.

Rheem I, Rheem S. Response surface analysis in the presence of the lack of fit of the second-order polynomial regression model. J Korean Data Anal Soc. 2012;14: 2995-3002.

Derringer G, Suich R. Simultaneous optimization of several response variables. J Qual Technol. 1980;12:214-219.