서 론
일반적으로 모유는 무균상태로 고려되어 왔다. 때문에 과학자들은 모유에서 발견되는 미생물이 시료채취 또는 저장과정에서 오염되었거나 유방염 등의 질병에 인한 것으로 생각했다. 이로 인해 자연스럽게 모유 내 병원성 세균을 검출하는 데 초점을 맞췄고, 이를 위해 배지를 이용한 배양 방법만을 사용해왔다. 당시의 관련 연구들은 모유 표본의 대부분이 무균 상태지만, 유방염에 걸린 여성의 모유가 Staphylococcus aureus를 주로 함유하며, 적은 양의 streptococci를 가진다고 보고했다(Thomsen et al., 1983, 1984; Osterman & Rahm, 2000). 그러나 유방염에 걸린 여성의 모유 표본에서 세균이 발견되지 않는 경우가 있었기 때문에 전문가들은 유방염이 감염성인지 비감염성인지, 유방염의 발병과 관련하여 세균의 정확한 역할은 무엇인지, 그리고 발병한 여성에게 항생제를 사용해야 하는 것인지에 대해 오랫동안 의문을 제기해 왔다(Fetherston, 2001; Kvist et al., 2008; Kvist, 2010). 보다 최근에는 분자적 기법을 사용함으로써 특정 DNA 단편의 변이를 기반으로 세균을 분류할 수 있게 되었다. 여러 연구진들은 분자적 기법을 통해 모유를 분석했고, 산모의 유방염 유무와 상관없이 모유에 풍부하고 다양한 세균 집단이 존재함을 밝혔다. 특히, 차세대 염기서열 분석법(Next Generation Sequencing, NGS)의 적용을 통해서 모유에 존재하는 세균의 전체적인 구성, 그 중에서도 배양기법에 의존적인 방법으로는 확인하기 어려운 혐기성 세균에 대한 전례 없는 식견들을 제공했다(McGuire & McGuire, 2015). 본 논문에서는 모유 세균총의 구성, 이들 구성에 영향을 주는 요인, 또 모유 내 세균의 기원을 밝히고자 한 최근의 연구 결과들을 소개하고자 한다.
본 론
건강한 여성의 모유가 가지는 세균 조성을 밝히기 위해 많은 연구진들이 분자적 기법을 이용하여 연구를 진행하였다(Table 1). 아마도 Martín 등(2003)의 연구가 첫번째일 것이다. 이들은 모유 수유 중인 8명의 건강한 스페인 여성과 이들의 아기를 통해 모유가 젖산균의 근원이라는 가설을 시험했다. 또한, 이들은 젖산균에 특이적인 배양기법과 분자적 기법인 Random Amplification of Polymorphic DNA(RAPD) 분석방법을 조합하였다. 그 결과, 모유, 유륜, 아기의 입, 아기의 분변, 모두에서 Lactobacillus gasseri와 Enterococcus faecium이 발견되었다. 모유에서 분리된 젖산균의 종류는 아기의 분변에서 분리된 것과는 동일했으나, 유방 피부에서 분리된 것과는 그 종류가 달랐다. 이를 근거로 연구자들은 모유가 아기에게 젖산균을 제공하는 중요한 공급원이 될 수 있으며, 이러한 세균이 단순히 주변 피부에서 오염된 것이 아니라 내생 기원일 것이라는 결론을 내렸다. 이후 Collado 등(2009)은 50명의 건강한 스페인 여성을 대상으로 모유 미생물총을 규정하기 위해 quantitative PCR(qPCR)을 진행하였다. 그 결과, 모든 표본에서 Bifidobacterium, Lactobacillus, Staphylococcus, Streptococcus가 분리되었고 Streptococcus가 가장 풍부했다. 해당 실험은 건강한 여성의 모유 미생물총을 규정하기 위해 qPCR을 이용한 첫 연구이며, 건강한 여성의 모유에 풍부한 세균 DNA가 존재함을 보고했다.
| Reference | Study population | Sequencing method | Major taxa identified | Conclusion |
|---|---|---|---|---|
| Martin et al., 2003 | - Spain | Cultured for LAB, RAPD analysis | LAB found in all milk samples, specifically Lactobacillus gasseri and Enterococcus faecium | HM can be an important source of LAB to the infant, and these bacteria have an endogenous origin. |
| - n=8 | ||||
| - 4 d postpartum | ||||
| Collado et al., 2009 | - Spain | qRTi-PCR | All samples contained Staphylococcus, Bifidobacterium, Lactobacillus, and Streptococcus | HM contains an abundance of bacterial DNA. |
| - n=50 | ||||
| Hunt et al., 2011 | - United States | Pyrosequencing | 9 core genera OTUs present in all samples: Staphylococcus, Streptococcus, Strratia, Pseudomonas, Corynebacterium, Ralstonia, Propionibacterium, Sphingomonas, Bradyrhizobioum | HM contains a diverse and complex bacterial community. |
| - n=16 | ||||
| - ~22-26 postpartum | ||||
| - 3 samples from each subject | ||||
| Cabrera-Rubio et al., 2012 | - Finland | Pyrosequencing, qPCR | Weisella, Leuconostoc, Staphylococcus, Streptococcus, and Lactococcus predominant in colostrum; Leuconostoc, Weisella, Lactococcus, and Staphylococcus predominant in mature milk | HM microbiome changes over lactation stages and differs by maternal weight and delivery mode. |
| -n=18 | ||||
| -0~2 d, 1 mo, and 6 mo postpartum | ||||
| Jost et al., 2013 | - Switzerland | Cultrues, Sanger sequencing, 454-pyrosequencing, 16S rRNA gene sequencing | Predominant: Staphylococcus, Streptococcus, and Propionibacterium | HM may significantly influence infant gut colonization and immune system. |
| -n=7 | ||||
| -3~6 d, 9~14 d, and 25~30 d postpartum | ||||
| Ward et al., 2013 | - Canada | Illumina sequencing | Predominant: Pseudomonas, Staphylococcus, and Streptococcus | Diversity of bacterial may be beneficial. |
| -n=1 (pooled milk from 10 women) | ||||
| -9~30 d postpartum | ||||
| Urbaniak et al., 2016 | - Canada | Illumina sequencing | Predominant: Firmicutes and Proteobacteria; at the genus level Staphylococcus, Enterobacteriaceae, Psedomonas followed by Streptococcus, Lactobacillus | HM contains a diverse population of bacteria. |
| -n=39 | ||||
| -6 d and onwards postpartum | ||||
| Williams et al., 2017 | - United States | High-throughput sequencing | Predominant: Firmicutes; at the genus level Streptococcus, Staphylococcus Relatively stable microbial communities within | HM produced by individual women. |
| -n=21 | ||||
| -2 d to 6 mo postpartum | ||||
| -9 samples from each subject | ||||
| Biagi et al., 2017 | - Italy | Illumina NGS | Predominant: Streptococcus, Staphylococcus, Bifidobacterium | HM may influence infant gut colonization and immune system. |
| -n=36 | ||||
| -20 d postpartum |
앞선 연구들을 기반으로 건강한 여성의 모유에 세균이 존재함을 증거하는 많은 연구가 발표되었다. 예를 들어, Hunt 등(2011)은 4주 동안 16명의 미국인 여성에게서 각각 3개의 모유 표본을 수집하고, 16S ribosomal RNA(rRNA) 유전자의 초가변 영역 V1-V2를 표적으로 하는 primer를 이용해 454-pyrosequencing을 진행함으로써 모유 미생물총을 특성화 했다. 속(genus) 수준에서의 생태계 분석 자료는 상당히 풍부하고 다양한 세균 군집을 나타냈지만, 모든 표본이 동일하게 보유한 세균 집단은 9개의 속(genus)뿐이었다. 저자는 이들을 “core”bacterial community라고 칭했으며, 여기엔 Staphylococcus, Streptococcus, Serratia, Pseudomonas, Corynebacterium, Ralstonia, Propionibacterium, Sphingomonas, Bradyrhizobium이 포함된다. 또한, 핵심 세균 집단은 전체 조작 분류 단위(operational taxonomic unit, OTU)의 52%를 차지하는 것으로 나타났다. 유사한 실험이 Cabrera-Rubio 등(2012)에 의해 수행되었다. 이들은 18명의 핀란드 여성에게서 얻은 모유의 미생물 군집을 세 시점(초유, 출산 후 1달째, 6달째)에서 분석하였다. 과(family), 속(genus) 수준에서의 분류 식별을 위해 16S rRNA 유전자의 초가변 영역 V1-V3를 표적으로 하는 primer를 사용하여 454-pyrosequencing과 qPCR 방법을 진행했다. 결과는 특히 초유에서 비교적 많은 양의 젖산균을 확인했고, Staphylococcus, Streptococcus, Veillonella, Leptotrichia, Prevotella 또한 높은 비율을 나타냈다. 그러나 해당 실험진은 Hunt 등(2011)이 보고한 것처럼 모유가 시기에 따라 독특한 미생물 구성을 보이는지는 밝히지 못하였다. 그러나 본인들이 밝혀낸 미생물 군집 구조가 Hunt 등(2011)이 밝힌 것과 큰 차이가 있음을 밝혔다. 이러한 결과적 차이는 실험을 진행한 표본집단의 차이와 실험 방법의 차이 때문에 발생했을 수 있다. 이를 확인하기 위해서는 추가적인 연구가 필요할 것이다.
Jost 등(2013)은 배양기법과 454-pyrosequencing 방법을 조합하여 모유의 미생물총을 분석한 연구 결과를 발표했다. 이들은 완전 모유수유 중인 7명의 건강한 스위스 여성에게서 출산 후 첫 한 달 동안 모유를 총 세 번 채취했다. Firmicutes, Proteobacteria 문(phylum)이 우세했고, 속(genus) 수준에서는 Staphylococcus, Streptococcus, Pseudomonas, Ralstonia, Bifidobacterium, Blautia, Bacteroides가 가장 풍부했다.
Ward 등(2013)은 수유 중인 10명의 여성에게서 출산 후 9∼30일 사이에 무균상태로 채취한 모유를 하나의 표본으로 합치고, 모유 표본이 가지는 모든 유전물질(metagenome)을 특성화하기 위해 Illumina Next Generation Sequencing(NGS) 기술을 사용했다. 모유에 있는 세균들은 문(phylum)과 속(genus) 수준에서 분류되었다. 모유의 미생물 군집 구조는 엄마와 아기의 분변 내 미생물 군집 구조와 문(phylum) 수준에서 비교하였다. 분석 결과, Proteobacteria와 Firmicutes 문(phylum)이 우세한 가운데 360개 이상의 속(genus)이 확인되었다. 속(genus) 수준에서는 Pseudomonas와 Staphylococcus가 전체 군집의 대다수를 차지했다.
최근에는 건강한 여성이 만든 모유가 가지는 미생물 군집의 조성에 관한 다양한 연구 결과들이 발표되고 있다. Urbaniak 등(2016)은 39명의 캐나다 여성을 대상으로 모유를 채취하고, 16S rRNA 유전자의 초가변 영역 V6를 표적으로 하는 primer를 이용하여 Illumina NGS 방법을 진행했다. 그 결과 Firmicutes, Proteobacteria 문(phylum)이 우세했다. 이는 Jost 등(2013)이 스위스에서 진행하여 454-pyrosequencing으로 분류한 결과와 동일하다. 속(genus) 수준에서는 Staphylococcus, Pseudomonas가 가장 풍부했으며, 그 다음으로 Sterptococcus, Lactobacillus 순으로 풍부했다. Williams 등(2017)은 수유 중인 21명의 건강한 미국인 여성을 대상으로 출산 후 2일부터 6달째까지 총 9번 모유를 채취했다. 표본들은 16S rRNA 유전자의 초가변 영역 V1-V2를 표적으로 하여 high-throughput sequencing을 통해 분류되었다. 모든 기간의 표본에서 Firmicutes 문(phylum)이 가장 우세했고, 속(genus) 수준에서는 Streptococcus와 Staphylococcus가 가장 우세했다. Biagi 등(2017)은 36명의 건강한 이탈리아 여성을 대상으로 모유를 채취하여 16S rRNA 유전자의 초가변 영역 V3-V4를 표적으로 하여 Illumina NGS 방법을 진행했다. 그 결과, Streptococcus, Staphylococcus, Bifidobacterium 속(genus)이 우세했다.
결과적으로 유방염 또는 다른 유방과 관련된 질병의 조짐이나 증상이 없는 건강한 여성이 만든 모유에 다양한 세균 군집이 존재한다는 것이 확실해졌다. 비록 연구집단에 따라 밝혀낸 세균 군집 조성에 차이가 있지만, 대부분의 연구가 Staphylococcus, Streptococcus 속(genus)을 함유한다고 보고했다(Fitzstevens et al., 2017). 그러나 연구들 간에 나타나는 차이가 어떤 요인으로 인해 발생한 것인지 추가적인 연구를 통해 밝혀내야 할 것이다(McGuire & McGuire, 2015).
앞서 소개한 여러 연구들처럼 최근 몇 년간 전문가들은 배지법과 분자생물학적 기법을 사용하여 모유에서 지속적으로 다양한 종류의 세균을 확인해왔다. 그러나 연구자들의 결과에 상당한 차이가 나타난다. 이러한 차이를 만드는 요인이 무엇인지 명확히 규정할 수는 없지만, 일부는 연구 간의 방법론적 차이에 의해 발생한 것으로 추정할 수 있다. 그 밖에도 인구학적, 환경적, 생리학적 변이들이 모유 미생물총에 영향을 줄 수 있다는 보고들이 있다. 구체적으로 지리적 위치, 수유 기간, 분만 형태, 산모의 비만, 산모의 건강(예; 복강 질병, HIV, 유방염), 의학적 치료(예; 화학적 치료)를 그 예로 들 수 있다(McGuire & McGuire, 2015). 또한 모유 미생물총은 모유에 존재하는 여러 구성요소에 의해서도 변화할 수 있다(Heikkilä et al., 2003; Kelsey et al., 2006; Bode, 2012; Hunt et al., 2012; Van Niekerk et al., 2014). Tušar 등(2014)에 의하면, 미생물 생태계는 한 여성의 두 유방 사이에서도 차이가 있을 수 있다.
아마도 가장 많이 보고된 것은 수유 기간과 분만 형태의 잠재적 영향에 관한 연구일 것이다. 예를 들어 Cabrera-Rubio 등(2012)은 핀란드 여성의 초유에 Weisella, Leuconostoc, Staphylococcus, Streptococcus, 그리고 Lactococcus 속(genus)이 우세한 반면, 성숙유에는 Veillonella, Leptotrichia, Prevotella가 더 많아졌다고 보고했다. 또한 제왕절개로 분만한 여성의 모유가 자연분만한 여성의 모유보다 높은 비율의 Acinetobacter를 가지는 등 분만 형태에 따라 모유 미생물총에 차이가 있음을 보고했다. 또한, Khodayar-Pardo 등(2014)은 32명의 스페인 여성을 대상으로 출산 후 첫 3주 동안 수유함에 따라 전체 세균의 수가 증가했고, 특히 Bifidobacterium, Enterococcus spp.의 수가 증가했음을 보고했다. 또한, 예정 출산일보다 일찍 출산한 여성보다 예정 출산일에 출산한 여성의 모유에서 Bifidobacterium spp.의 농도가 항상 높았고, 자연분만한 여성보다 제왕절개로 분만한 여성의 초유가 더 높은 세균 농도를 나타냈다. 또한, Bifidobacterium spp.는 제왕절개로 분만한 여성보다 자연분만한 여성의 모유에서 더 빈번하게 검출되었다. 해당 연구는 Cabrera-Rubio 등(2012)의 연구와는 다르게 NGS 방법이 아닌 표적 PCR 분석 방법을 사용했기 때문에 전체적인 미생물 구성의 변화는 확인할 수 없었다.
그러나 Urbaniak 등(2016)은 모유 미생물총이 분만 형태에 영향을 받지 않는다고 발표했다. 이들은 Cabrera-Rubio 등(2012)이 비만인 여성과 그렇지 않은 여성의 모유를 채취하여 진행한 연구임에도 각 집단의 비율을 밝히지 않았고, 산모의 비만과 분만 형태가 미치는 영향을 같은 표본을 가지고 분석하였으며, 통계적 분석 없이 막대그래프 상의 작은 차이를 가지고 분만 형태에 따른 미생물총의 차이로 보았기 때문에 그 결과를 믿을 수 없다고 주장했다. 또한 심각하게 조산한 산모의 모유도 출산 예정일에 출산한 산모의 모유와 그 구성이 크게 다르지 않았다고 밝혔다. Williams 등(2017)은 분만 형태에 따른 모유 미생물총의 차이는 확인했으나, 수유 기간과 관련해서는 한 여성의 모유 미생물총이 비교적 일정하게 유지된다고 보고했다. 시간이 지남에 따라 소수의 세균 집단이 증가하기는 했으나, 가장 풍부한 10개 속(genus)의 경우 그 조성의 변화가 미미했다. 이러한 결과적 차이의 정확한 원인은 알 수 없으나, 채취 방법, 분석 방법의 차이를 원인으로 고려할 수 있다.
Cabrera-Rubio 등(2012)은 또한 Body Mass Index(BMI)가 30 이상인 비만 산모의 모유와 BMI 25 이하인 정상 체중 산모의 모유를 비교했을 때 미생물의 구성이 서로 다르고, 비만 산모의 모유가 세균 다양성이 적다고 보고했다. 표본 집단의 식단 기록이 없기 때문에 에너지 균형, 미생물 영양상태 등 전체적인 영양상태에 따른 효과는 확인할 수 없었으나, 같은 연구실에서 유사한 방법으로 채취, 분석되었기 때문에 저자는 두 집단의 차이가 채취 방법이나 실험 방법에 의한 것이 아니라, BMI의 차이에 의한 것이라고 결론지었다. 또한, Collado 등(2012)은 산모의 비만, 산후 기간, 그리고 모유의 미생물총 사이의 잠재적 상관관계를 확인하기 위한 연구를 진행했다. 56명의 핀란드 여성(22명은 과체중, 34명은 정상체중)을 대상으로 출산 후 1∼2일, 1달, 그리고 6달째에 모유를 채취하여 qPCR을 진행했다. 분석 결과, 정상체중의 산모와 비교했을 때 과체중 산모의 모유에 Staphylococcus 속(genus) 세균이 더 많았고, Bifidobacterium 속(genus) 세균이 더 적었다. Williams 등(2017)도 산모의 비만이 모유 미생물총의 문(phylum) 수준에는 영향을 주지 않지만 속(genus) 수준에서 Granulicatella의 증가와 관련이 있었다고 보고했다.
몇몇의 연구 집단들은 모유 내 미생물총과 관련하여 산모의 건강 상태에 초점을 맞췄다. 예를 들어 González 등(2013)은 에이즈 양성반응을 보이는 아프리카 여성의 모유가 높은 세균 다양성과 높은 Lactobacillus spp. 빈도를 보였으며, 에이즈 양성반응을 보이는 여성의 모유에는 Staphylococcus hominis와 S. aureus가 낮은 빈도로 발견됨을 밝혔다. 서로 다른 미생물 생태계와 관련된 여러 요인들이 에이즈 감염에 대한 민감성에 영향을 주었을 수 있다. 그러나 에이즈 감염으로 인해 미생물 군집이 변화한 것일 수도 있다. 또한, Olivares 등(2014)은 심장병이 있는 12명의 스페인 여성과 12명의 건강한 스페인 여성을 대상으로 실험을 진행했다. 이들은 심장병이 있는 여성보다 건강한 여성의 모유에 Bifidobacterium spp.와 Bacteroides fragilis의 수가 더 많았다고 보고했다. 이러한 변화는 TGF-b1, 분비성 immunoglobulin A와 같은 모유 내 면역보호 물질의 농도 감소를 동반했다. 또한 Grönlund 등(2007)은 알레르기가 없는 여성에 비해 알레르기가 있는 여성의 모유에서 Bifidobacterium이 더 적게 발견되었다고 밝혔다. 최근에는 Jiménez 등(2015)이 건강한 10명의 산모와 유방염이 있는 10명의 산모를 대상으로 모유 내 metagenome을 비교했다. 건강한 산모의 모유와는 다르게 급성 유방염에 걸린 산모의 경우 Staphylococcus aureus가 명백히 높은 비율을 나타냈고, 아급성 유방염의 경우 Staphylococcus epidermidis 관련 유전자가 증가했다. 이러한 연구들이 보고한 차이들이 오로지 질병에 의한 것인지, 아니면 다른 요인에 의한 것인지 확실히 하기 위해서는 동물 모델을 동반한 대규모 임상 실험을 진행할 필요가 있을 것이다(McGuire & McGuire, 2015).
앞서 언급한 인류학적, 환경적, 생리학적 변이에 더하여 모유 내 구성 성분 또한 세균 군집에 영향을 주거나, 세균에 의해 영향을 받을 수 있다. 이러한 관점에 가장 부합하는 모유 성분은 human milk oligosaccharides(HMO)일 것이다(Bode, 2012). HMO는 섭취한 아기의 소화관에서 유익균의 성장을 촉진시킬 수 있기 때문에 천연의 “prebiotics”로 알려져 있다. Hunt 등(2012)은 HMO가 모유에 존재하는 미생물 군집과 상호작용할 수 있음을 시험관 상에서 증명했고, 특히 Staphylococcus spp.의 생장과 관련이 있었다. 또한, conjugated linoleic acid와 같이 모유에 존재하는 다양한 지방산이 모유 내 세균의 생장을 조절할 수 있다는 것도 확인되었다(Kelsey et al., 2006). 뿐만 아니라 Heikkilä와 Saris(2003)가 밝힌 것처럼 모유 내 미생물 간의 상호작용 또한 일어날 수 있다.
모유 내 미생물의 기원이 어디인가 하는 것도 뜨거운 논쟁 거리 중 하나이다. 이와 관련한 연구 결과들은 이들의 기원이 복잡하다고 말하고 있다. 예를 들어, 모유 수유를 하지 않은 산모의 유선조직에 살아있는 세균이 존재한다고 보고된 바 있다(Latuga et al., 2014; Xuan et al., 2014; Chan et al., 2016). 이러한 발견은 유선 자체가 모유에 세균을 제공하는 하나의 공급원이 될 수 있음을 강력히 시사한다(Rodríguez, 2014, McGuire & McGuire, 2017). 모유에 존재하는 미생물들이 어떻게 유입된 것인지 확실하지 않지만 크게 두 가지 경로로 추측하고 있다. 유선관을 통해 외부로부터 유입되거나, entero-mammary pathway를 통해 소화관에서 기인하거나, 두 경로 모두를 통해 유입될 수 있을 것이다(Fernándeza et al., 2013; Jost et al., 2015; Fig. 1).
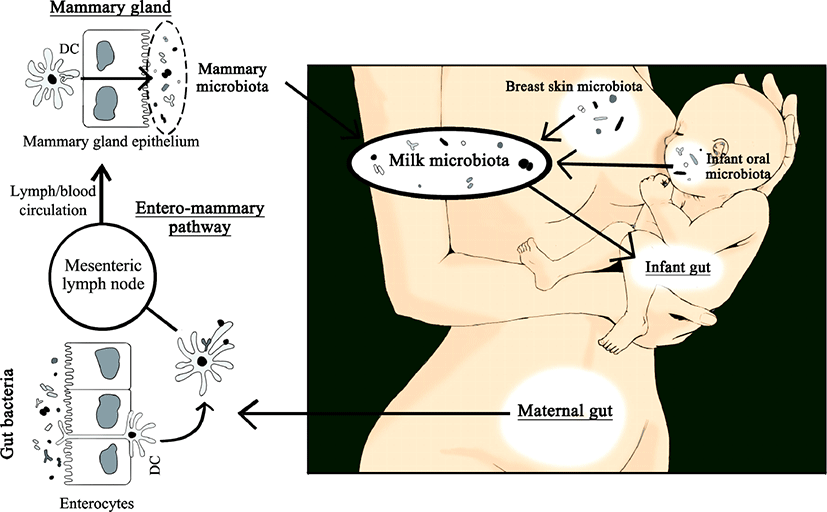
사람의 각 신체부위에 존재하는 미생물들은 서로 밀접한 관계를 보이며 활발하게 상호작용한다(Costello et al., 2009). 따라서 모유의 미생물총 또한 산모 그리고 아기와 관련된 다른 미생물 군집에 노출됨으로써 지속적으로 영향을 주고받을 수 있다. Ramsay 등(2004)은 모유를 수유하는 동안 모유가 다시 유선관으로 역류할 수 있다고 밝혔다. 이러한 과정은 아기의 입과 유선 사이의 세균 교환을 가능하게 하는 이상적인 경로가 될 수 있다. 사람의 타액 내 미생물총과 관련하여 아직까지 알지 못하는 부분이 더 많지만, 성인과 치아가 나지 않은 아기의 침 모두에서 Streptococcus spp.가 우세한 것으로 나타났다(Aas et al., 2005; Cephas et al., 2011; Yang et al., 2012). 또한 Streptococcus spp.는 초유와 성숙유 모두에서 풍부하게 존재하는 계통형 중 하나이다(Jiménez et al., 2008a, 2008b; Hunt et al., 2011). 이는 모유의 미생물총이 침의 미생물총 성립에 중요한 역할을 할 수 있다는 가설을 뒷받침한다. 그러나 이는 아기를 낳기 전의 산모에게서 나오는 전초유가 이미 미생물을 가지고 있다는 결과를 설명하지 못한다(Martín et al., 2004).
Staphylococcus, Corynebacterium, Propionibacterium과 같이 성인 피부에 일반적으로 존재하는 일부 세균 계통형들이 모유에서도 빈번하게 발견되었다(Gao et al., 2007; Grice et al., 2009; Gao et al., 2010). 이는 산모의 피부 미생물총과 상호작용함으로써 모유의 미생물총 구성에 영향을 줄 수 있다는 가능성을 시사한다. 그러나 모유 내 세균 군집 구조와 유방 피부의 세균 군집 구조를 비교했을 때 일부 계통형을 공유하고 있으나, 주요 차이점 또한 존재했다(Hunt et al., 2011). 두 군집 간의 이러한 차이들은 모유 미생물총이 단순히 피부로부터 오염된 것이 아님을 나타낸다. 또한, 독립적인 두 연구가 lactobacilli 또는 bifidobacteria가 산모의 모유 표본에서는 분리되었으나 유방 피부 샘플에서는 분리되지 않을 수 있음을 보였다(Gueimonde et al., 2007; Martín et al., 2009). 보다 전에 발표된 Martín 등(2003)의 연구에서는 모유에서 분리한 lactobacilli와 enterococci가 같은 개체의 피부에서 분리한 lactobacilli, enterococci와 유전형적으로 다르게 나타났다. Bifidobacteria의 경우, 이들은 절대 혐기성 미생물에 속하기 때문에 근본적으로 피부는 이들의 기원이 될 수 없다(Gueimonde et al., 2007). 이와 같은 연구 결과들은 적어도 모유 미생물총의 일부는 내인성 경로를 통해 산모의 장에서 기인했을 것이라는 제안을 뒷받침한다.
비록 어떻게 세균이 장상피를 통과하여 유선으로 이동할 수 있는지, 그 경로나 기작을 아직은 완벽히 규명하지 못했지만, 몇몇의 연구들이 그럴듯한 과학적 근거를 제공했다. Bertotto 등(1991)이 항원에 의해 활성화된 세포들이 림프조직계를 순환함으로써 유선으로 이동할 수 있음을 보였고, Vazquez-Torres 등(1999)과 Rescigno 등(2001)은 각각 수상돌기세포와 대식세포가 장상피를 통과할 수 있음을 확인했다. 또한, Macpherson 등(2004)이 수상돌기세포과 대식세포가 살아있는 세균세포를 며칠간 보유할 수 있음을 밝혔다. 이러한 결과들을 근거로 모유에서 발견되는 살아있는 세균의 기원이 산모의 장일 수 있으며, 이들 세균이 산모의 수상돌기세포와 대식세포가 관여하는 내인성 경로를 통해 유선으로 도달했을 것이라는 가설을 세울 수 있었다(Fernández et al., 2013). 이후 이를 뒷받침하는 여러 연구들이 보고되었으나(Langa, 2006; Perez et al., 2007), 아직은 더 많은 연구가 필요한 단계이다.
결 론
건강한 여성의 모유가 다양하고 풍부한 미생물 군집 구조를 가지고 있음이 확실하다. 뿐만 아니라 표본집단의 국적, 모유 채취방법, 미생물 분석 방법이 서로 다름에도 불구하고, 서로 다른 연구결과들이 공통적으로 Streptococcus와 Staphylococcus 속(genus)을 모유 미생물총의 구성원으로 제시하고 있다. 이를 근거로 했을 때, 아마도 Streptococcus와 Staphylococcus는 모유가 일반적으로 가지는 세균 속(genus)인 것으로 보인다. 수유 기간, 분만 형태, 산모의 비만, 산모의 건강, 화학적 치료 여부 등의 요인으로 인해 모유 미생물총이 영향을 받을 수 있다는 연구 결과들이 있으나, 이를 반박하는 연구 결과 또한 존재하기 때문에 모유 미생물총에 영향을 주는 요인을 정확히 밝히기 위해서는 더 많은 연구가 필요할 것이다. 모유 내 미생물의 유입 경로는 크게 유선관을 통한 외인성 경로와 entero-mammary pathway라는 내인성 경로로 생각되고 있다. 모유 미생물총의 기원에 관해서는 아직 더 많은 연구가 필요한 상태지만, 지금까지의 연구 결과를 근거로 했을 때 모유 내 미생물들이 다양한 장소에서 기인했음은 분명하다. 모유는 아기가 일정 기간동안 유일하게 먹는 식품으로 여기에 미생물이 존재한다면 반드시 그 역할이 있을 것이다. 전문가들은 모유 내 미생물이 “probiotics”로 작용하여 아기의 초기 장내 미생물총 형성에 도움을 줄 것으로 추측하고 있다. 따라서 모유 미생물총의 존재에 대한 발견을 넘어 모유가 가지는 세균의 특성과 이들이 아기의 건강에 미치는 영향에 대한 연구가 활발히 진행되어야 할 것이며, 이러한 연구들은 모유에서 분리한 균주를 이용하여 probiotics의 개발을 가능하게 하고, 이를 적용하여 보다 모유에 가까운 조제분유를 만들 수 있게 할 것으로 기대된다.






