서 론
현재 Probiotic 연구는 새로운 도전에 직면하고 있는데, 이것은 미국과 유럽에서 이전보다 강화된 관련 법규의 적용으로 probiotic의 건강에 대한 연구를 엄격히 제한하고 있기 때문이다(Papadimitriou et al., 2015). 물론 이와 같은 엄격한 규제가 주요 문제는 아닐 수 있지만, 하지만 이것은 새로운 probiotic 균주 및 응용에 상당히 방해하는 요소로 작용할 수 있을 것이다. 왜냐하면, 현재 건강을 유지하고 복원하는 능력 연구에 있어서 미생물의 조작방법으로 많은 새롭고 흥미로운 작용가설이론을 만들 수 있는 metagenomics 연구와 노력이 활발하게 진행되고 있기 때문이다. 우선, 이 metagenomics 연구 결과는 수많은 균주들을 검사하는데 기존의 방식보다 매우 효율적으로 진행할 수 있으며, 또한 다양한 건강 상태를 가진 사람들(예를 들면, 저체중 과비만, 알레르기와 비알레르기 등)과 비교하여 가능성이 있는 새로운 건강증진 세균들을 식별할 수 있다. 여러 가지 새로운 응용 프로그램은 보충 음식물 또는 약품을 통해서 이미 제안되어지고 있다. 또한 Faecalibacterium prausnitzii 수준의 향상은 염증성 장질환(IBD)의 환자에 유익으로 제안되어 있으며, Akkermansia muciniphila의 사용은 대사장애를 치료할 수 있다고 최근에 특허로 등록되었으며, 자폐증범주성장애(autism spectrum disorders)에 있어서 식이 생리활성 단백질과 펩타이드의 역할은 새로운 probiotic 균주로서 가능성을 가지고 있다(Miquel et al., 2013; Siniscalco and Antonucci, 2013; Cani et al., 2014; Papadimitriou et al., 2015). 오늘날 법규 체계는 이러한 새로운 것들을 적용하는데 능동적으로 대처할 준비가 되지 있지는 않다. 일반적으로 승인을 얻기 위해서는 작용 기전의 완벽한 분석을 요구한다. 즉, 위험의 모든 요소들은 다양한 집단에서, 다양한 용량으로 그리고 다양한 운반형식과 다양한 매트릭스를 사용하였을 때 결정될 것이다. 본 총설논문에서 제시된 연구 방법들은 이러한 과정 등을 자세하게 소개하는 것도 목적의 일부분이기도 하다.
여기에 설명된 많은 in vitro 방법은 비록 구식으로 생각될지라도, 여전히 비용면에서 저렴하고 윤리적으로 안전하기 때문에 아직도 사용되고 있는 방법들이다. 그리고 새로운 분자 omics 기반 기술의 사용은 빠르게 널리 이용되고 있는데, 이것은 머지않아 전통적인 기존의 검사 방법을 대체할 수 있을 것으로 예상된다(Papadimitriou et al., 2015). 또한 Omics 기술은 현재의 방법인 in vitro 또는 in vivo 검사 결과로부터 얻어진 probiotic 후보 균주들의 후속 분석에서도 매우 효과적으로 역할을 할 것이다. 예를 들어, 유전체 서열은 항생제 내성 또는 병원성 유전자의 존재를 파악하여 잠재적인 위험을 일으킬 수 있는 균주를 신속하게 발견하고 제거할 수 있기 때문이다. 또한 새로운 연구 방법은 건강 기능성 강조표시(health claim) 또는 약품관련 서류의 작성을 용이하게 하며, 또한 기능성 작용기전의 분석과 설명을 가능하게 할 수 있다. 무엇보다 중요한 것은 이 기술을 사용하면 더 이상 살아있는 미생물이 필요하지 않다는 것인데, 이것은 그들은 활성 화합물로 확인된 활성성분 또는 대사산물에 의해서 대체될 수 있기 때문이다. 이것은 식품에서 약품제조 영역까지 특정 분야에 응용할 수 있게 될 것이다. 하지만 현재의 probiotic 연구에서 예상되는 것처럼, 어떠한 부작용이 없는 활성 성분으로 변화되는 것이 먼저 밝혀져야 한다는 것이다. 따라서 본 총설논문에서 probiotic 연구에 이용되는 다양한 모델과 연구방법들이 설명되고, 각 방법들의 장점과 한계점 그리고 해결책과 향후 전망이 자세하게 서술되어 있다. 또한 본 총설논문의 모든 자료들은 이미 발표된 다양한 문헌 등을 재정리하여 서술하였다.
Probiotic 연구에 현재 이용되고 있는 In Vitro, In Vivo, Omics의 이용에 대한 연구 현황
Probiotic 연구의 초기부터 in vitro 검사는 매우 단순하지만 비용면에서 비교적 저렴하기 때문에 가장 선호하는 방법이다. 비록 이러한 검사방법들 가운데 일부는 시대에 뒤떨어진 것처럼 보일 수도 있지만, 이것들은 여전히 사용되고 있으며, 심지어 최근의 연구보고서에도 자주 이용되고 있는 것은 사실이다(Papadimitriou et al., 2015). 무엇보다 in vitro 검사의 가장 중요한 장점은 여러 균주들을 동시에 선별할 수 있는 능력일 것이다. 더 자세한 연구내용은 Table 1에 자세하게 정리되어 있다.
| The most recently published articles | The test for screening | The property of probiotics |
|---|---|---|
| Hughes and Hoover (1995) | Β-Galactosidase activity | Additional health benefits |
| Pompei et al. (2007) | Production of vitamis | |
| Kullisaar et al. (2002) | Linolenic acid test | |
| Campieri et al. (2001) | Oxalate-degradation | |
| Burns and Rowland (2004) | Ames test | Anticancer |
| Pool-Zobel et al. (1996) | Comet assay | |
| Duangjitcharoen et al. (2014) | Nitrosamine degrading assay | |
| Choi et al. (2006) | Preventing colon cancer cell invasion | |
| Castro et al. (2010) | Induction of apoptosis of cancer cells | |
| Faridnial et al. (2010) | Binding to mutagenic compounds | |
| Nybom et al. (2009) | Removal of toxins and toxic metals | |
| Cousin et al. (2012) | Bacterial fermentation and production of SCFAs | |
| Coman et al. (2014) | Production of antimicrobial metabolism such as organic acids and bacteriocins | Antimicrobial assays |
| Bao et al. (2010) | Co-aggregation with pathogens | |
| Ewaschuk et al. (2008) | Enhancement of intestinal barrier function | |
| Zheng et al. (2013) | Deconjuation of bile salts | Cardiovasular diseases |
| Lye et al. (2010) | Conversion of cholesterol to coprostanol | |
| Papadimitriou et al. (2007) | Peptides from bacterial metabolism with ACE inhibitory activity | |
| Garcia-Cayuela et al. (2014) | Cell surface hydrophobicity | Colonization of the host |
| Kinoshita et al. (2013) | Adhesion to mucus | |
| Botta et al. (2014) | Auto-aggregation screening | |
| Tassell and Miller (2011) | Adhesion to intestinal epithelium | |
| Corthesy et al. (2007) | Bacterial translocation in the GT | Immunomodulation |
| Cencic and Langerholc (2010) | Co-culture models mimicking in vivo situation | |
| Steinberg et al. (2014) | Interaction of host immune system with bacterial compounds | |
| McKay et al. (1997) | Regulation of epithelial tight junctions | |
| Foligne et al. (2007) | Anti-inflammatory immune-stimulating properties | |
| Kim et al. (2013) | β-hexosaminidase release assay | |
| Pisano et al. (2014) | Antibiotic resistance | Safety assays |
| Pisano et al. (2014) | Hemolytic activity | |
| Harty et al. (1994) | Adhesion to mammalian cells | |
| Bernardeau et al. (2006) | Production of enzymes | |
| Tan et al. (2013) | Production of toxins | |
| Bover-Cid and Holzapfei (1999) | Production of biogenic amines | |
| Van den Abbeele et al. (2012) | Low pH and bile | Survival stress within the host |
Source: Papadimitriou et al., 2015
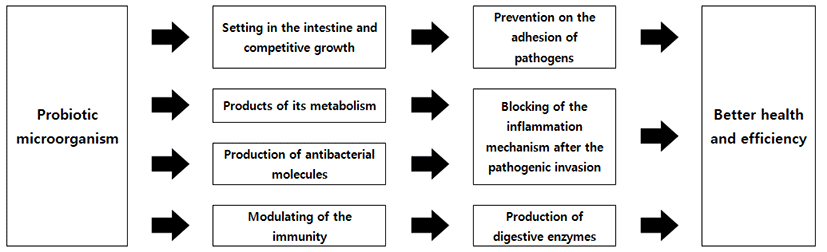
잠재력을 가지고 있는 Probiotic을 탐구하기 위해서 in vivo 방법의 이용은 다양하게 복잡한 거대한 생물학적 모델로 설명되어질 수 있을 것이다(Fig. 1). 여기에는 매우 간단한 다세포 생물에서 시작하여 벌레, 무척추동물, 정교하게 knock-out 모델을 적용한 설치류 그리고 최종적으로 다양한 인종별 인간 임상실험까지 넓은 범위가 속한다(Papadimitriou et al., 2015). 여기에 속한 모델들은 귀중한 정보를 제공하기도 하지만, 한 가지 단점으로는 다른 모델의 결과를 통합하는 것은 매우 어려운 숙제로 남아 있다는 것이다. 따라서, probiotic 기능의 최종 평가는 목표대상에서 직접 수행되는 것이 가장 이상적이지만, 하지만 균주들의 예비선택은 가장 적합한 in vivo 모델을 사용하여 고가의 임상 실험이 포함되어야 한다는 것이다(Papadimitriou et al., 2015). 더 자세한 연구내용은 Table 2에 자세하게 정리되어 있다.
| The most recently published articles | The screening of intervention | The functionality of probiotics |
|---|---|---|
| Sodhi et al. (2012) | Tissue-specific knock-out | Closely related innate and adaptive immunity |
| Martin et al. (2008) | Humanized mice | Hosting complex microbiota |
| Verdu and Collins (2004) | Axenic mice | |
| Eaton et al. (2011) | Monocolonized mice | |
| Le Roy et al. (2013) | Microbiota transplantation | |
| Henao-Mejia et al. (2012) | Co-housing | |
| Viaud et al. (2013) | Selective antibiotic treatment | |
| Helm and Burks (2002) | Transgenic mice | Physiological relevance for humans |
| Kim et al. (2014) | Allergy, inflammation | Responsiveness to many infectious, immune and other disorders |
| Kikuchi et al. (2014) | Bacteria, virus, fungi and parasites pathogens | |
| Kwon et al. (2013) | Neurologic disorders | |
| Hsiao et al. (2013) | Stress, cognitive functions | |
| Sodhi et al. (2013) | Conditional knock-out | Sharing of similar immune response types |
Source: Papadimitriou et al., 2015
지난 20년 동안 분자표지인자를 식별하기 위해서 다양한 시도들이 진행되었으며, 특히 probiotic 균주들의 선별에 적용되어서 신속하면서도 정확하게 확인할 수 있게 되었다. 예를 들면, 스트레스와 같은 악조건에서 probiotic 미생물들의 복잡한 적응 메커니즘을 설명하기 위해서, 여러 가지 유전자 및 omics 연구들은 스트레스와 관련된 유전자 발현 및 단백질 생산 형태를 식별하기 위해서 시도가 진행되었다(Papadimitriou et al., 2015). 더 자세한 연구내용은 Table 3에 자세하게 정리되어 있다.
| The most recently published articles | The screening of gene/protein | The function of probiotics | |
|---|---|---|---|
| Turpin et al. (2012) | Adhesion | Mub | Cell-surface proteins with cell wall |
| Turpin et al. (2012) | slpA | S-layer protein | |
| Turpin et al. (2012) | Apf | Aggregation promoting factor | |
| Munoz-Provencio et al. (2012) | srtA | Sortase-dependent surface protein | |
| Turroni et al. (2013) | spaCBA, spaFED, pil2, pil3, fim1, fim2, fim3 | Sortase-dependent biosynthesis of pili | |
| Vastano et al. (2014) | FbpA, E1 β-subunit of the pyruvate dehydrogenase complex | Fibronectin binding protein | |
| Westermann et al. (2012) | tad | Assembly of tide adherence pilus | |
| Sela et al. (2008) | Degradation of HMOs and Mucus | 43 kbp gene cluster | Catabolism of HMOs |
| Sela et al. (2008) | F1SBPs | Import of oligosaccharides | |
| Yoshida et al. (2012) | β-Galactosidases | Degradations of type-1 and type-2 HMOs | |
| Kim et al. (2009) | Glycosylases | Degradation of HMOs | |
| Shimada et al. (2015) | Glycosyl hydrolases, exo-α-sialidases, fucosidaes, PTS systems, ABC-type carriers, specific permeases, engBF, afcA, NagBb, agnB | Mucin degradation | |
| Mack et al. (2003) | adh | Adhesion and stimulation of mucin secretion | |
| Wang et al. (2014) | Soluble protein p40 | Stimulation of mucin production | |
| Bauerl et al. (2010) | Modulation of the immune system | P40 and p75 proteins and homologues | Activation Akt, promotion of cell growth, inhibition of TNF-α |
| Gilad et al. (2011) | ClpB, Rpf | Potential immunogenic proteins | |
| Le Marechal et al. (2014) | SLPs, additional disperse genetic loci | Regulation of anti- or pro-inflammatory immune responses | |
| Turroni et al. (2010b) | ser | Inhibition of elastases | |
| Fanning et al. (2012) | Cell surface-associated EPS | Adaptive immune response and protection against the gut pathogen Citrobacterrodentium | |
| Schlee et al. (2007) | Flagellin | Induction of human β-defensin 2 | |
| Corr et al. (2007) | Production of antimicrobial compounds | Bacteriocins | Induction against enterophogens |
| Meijerink et al. (2010) | Genes involved in plantaricin biosynthesis and secretion | Regulation of pro- and anti-inflammatory cytokines of DCs | |
| Saulnier et al. (2011) | Production of nutrients and other beneficial processes | Vitamins, essential amino acids, SCFAs | In situ production of important nutrients |
| Kim et al. (2009) | fos | Processing of health-promoting fructooligosaccharides | |
| Fukuda et al. (2012) | ABC carbohydrate transporters | High production of acetate and protection from enteropathogenic infection | |
| Lee et al. (2010) | ccpA | Influencing blood cholesterol | |
| Jacobi et al. (2012) | Quorum sensing | luxS | Induction of anti-inflammatory cytokines |
| Moslehi-Jenabian et al. (2011) | luxS | Adhesion and competitive exclusion of pathogens | |
| Fujii et al. (2008) | lamBDCA operon, lamKR operon | agr-like quorum sensing systems | |
| Mitsuma et al. (2008) | Quorum sensing system related peptide | Induction of c-myc and IL-6 genes in somatic cells | |
| Hamon et al. (2014) | Stress responses (acid and bile) | Heat shock proteins | Repair of damaged proteins |
| Hamon et al. (2014) | Clp proteases | Refolding or degrading denatured proteins | |
| An et al. (2014) | uvrB, uvrD1, vsr, helicases | DNA repari | |
| Koponen et al. (2012) | F1F0-ATPase | Decreases of intracellular pH | |
| Jin et al. (2012) | manB, glum, dapA, glycosyltransferases | Peptidoglycan biosynthesis | |
| Koskenniemi et al. (2011) | dit operon | D-alanylation of lipoteichoic acids | |
| An et al. (2014) | fab genes | Fatty acid biosynthesis | |
| Jin et al. (2012) | Etk-like tyrosine kinase, welG | Exopolysaccharide biosynthesis | |
| Hamon et al. (2014) | FabF, RfbB, RfbC | Cell envelope biogenesis | |
| Koponen et al. (2012) | luxS | Cell-to-cell communication | |
| An et al. (2014) | bsh | Deconjugation of bile salts | |
| An et al. (2014) | Transporters | Bile efflux | |
Source: Papadimitriou et al., 2015
Probiotic 연구에 있어서 In Vitro, In Vivo, Omics의 이용에 대한 향후 전망
건강 기능성 강조표시(health claim)와 연관되어 probiotic 성질을 미리 결정하거나 또는 문서화에 이용될 수 있는 다양한 in vitro 방법들이 존재한다. 비록 이와 같은 분석은 probiotic 후보균들을 선별하는데 매우 유용하지만, 그들은 매우 다양한 효과를 나타내고 있다는 것이다(Papadimitriou et al., 2015). 따라서 관련 기관들은 상세한 실험방법과 지침을 문서화하여 in vitro 검사의 표준화를 시도하고 있다. 하지만, 현실적으로 in vitro 검사가 다분히 임의의 방식으로 수행되고 있음이 관련 문헌조사에서 밝혀졌다. 따라서 이런 것들이 다른 연구자들에 의해서 진행된 연구결과를 상호 비교하는 것을 어렵게 만드는 것이다. 더 나아가서 probiotic 균주의 선별을 위한 in vitro 검사의 결과를 전적으로 의존하는 것을 어렵게 만드는 것은 바로 재현성 문제 때문이라고 보고되었다(Papadimitriou et al., 2015). 물론, in vivo 분석이 매우 적합하지만, 대부분의 경우에서 비용과 윤리적인 문제 때문에서 고속처리탐속기술(high throughput screening)을 사용할 수가 없기 때문이다. 따라서, in vitro 검사는 새로운 probiotic 균주의 발견에 있어서 매우 중요하며, 필요한 부분으로 여전히 인식되고 있다. 향후 추가 연구에서는 반드시 2가지의 것이 고려되어야 하는데, 첫째는 probiotic 잠재능력을 가진 균주들의 재현성 향상이며, 둘째는 의양성(false positive) 또는 위음성(false negative)의 비율을 낮추는 것이다. 이러한 목적으로 가능하게 이용될 수 있는 실험 방법을 개발하고 표준화시켜야 할 것이다(Papadimitriou et al., 2015). 또한 현재의 in vitro 시험의 평가로도 건강증진 특성을 확대할 수 있는 새로운 방법이 반드시 개선되거나 또는 개발되어야 할 것이다.
동물 모델에 있어서 정확성을 증가시키기 위해서는 인간 유전자의 기능을 가진 세포, 조직 또는 기관 등을 가지고 있는 마우스(multi- humanized mice)가 고려되어질 수 있다. 인간 세포 또는 조직의 수용체로서 면역결핍 마우스(immune-deficient mice)가 종종 사용되고 있는데, 왜냐하면 그들은 숙주 면역의 결핍 때문에 이종 세포를 수용하는 것이 상대적으로 쉽게 때문이다(Papadimitriou et al., 2015). 전통적으로는 이러한 목적을 위해서는 누드 마우스(nude mice) 및 중증 합병 면역 결핍증(SCID) 마우스가 주로 사용되었는데, 하지만 더 효율적으로 인간의 세포와 조직을 생착할 수 있는 다른 많은 모델들도 존재하고 있다(Ito et al., 2008). 이러한 인간화된 마우스 모델은 인간의 건강과 질환에 있어서 다양한 시나리오에 있어서 인간의 면역 시스템을 모델링하는데 도움을 줄 수 있으며, 또한 인간의 생리에 더 가깝게 설정된 in vivo에서 치료용으로 사용된 다양한 후보들의 평가를 가능하게 할 수 있다는 장점이 있다. 하지만 이렇게 특수하게 인간화된 마우스는 인간 치료를 위해서 생물학적 그리고 의약적인 연구에 있어서 일반적으로 사용되고 있지만, 아직까지는 probiotic 연구에 있어서 자주 이용되고 있는 것은 아니다(Papadimitriou et al., 2015).
숙주의 많은 면역 및 대사 기능을 위한 미생물 균총의 중요성을 감안하여, 인공적으로 만들어진 미생물 균총을 가진 마우스 모델의 개발 및 사용(예를 들면, 인간 미생물균총)은 더 나은 인간 조건을 만들어서 연구하는데 많은 도움을 줄 수 있을 것이다. 무균(axenic) 또는 한정성(monoxenic) 마우스의 사용은 위에서 언급한 것처럼 “계란 또는 닭”에 질문을 해명하는 데 도움을 줄 수 있을 것으로 사료된다. 미생물 균총의 유무에 관계없이 식이 개입의 영향은 간접적인 대사 또는 미생물학적인 효과와 대비하여 섭취된 probiotic의 직접적인 영향에 대한 흥미로운 것들을 알 수 있으며, 또는 어떤 계획된 식이개입의 면역체계에 있어서도 직접적으로 영향을 나타낼 수도 있다.
물론 동물 모델을 사용할 경우, 윤리적 문제가 논란이 되고 있는 것은 사실이다. 하지만 최근 네덜란드의 Wageningen 대학에서 이 문제에 대한 해결책으로 흥미로운 결과를 보고하였는데, 이것이 향후 대안으로 이용될 수 있을 것으로 사료된다(Papadimitriou et al., 2015). 이것은 바로 컴퓨터 시뮬레이션과 같은 가상 환경에서의 방법(in silico solution)으로서 존재하는 미생물과 상피세포와 돼지 창자, 영양소, 다양한 식품(또는 사료)과의 상호 작용을 설명하는데 사용될 수 있다는 것이다. 수학적 모형에서는 모든 요소들은 상호 양적 종속성을 가진 노드(node)로 간주되어진다. 이러한 모델의 상호작용을 이용하여서 장내 면역, 저항력과 장벽 기능과 연관되어서 높은 수준의 처리공정은 시뮬레이션되어질 수 있으며, 장 항상성(homeostasis)과 같은 조건들도 더 잘 이해될 수 있을 것이다(Papadimitriou et al., 2015). 장기적인 관점에서 이러한 모델은 가설을 검증할 수 있을 정도로까지 역동적이고 예측 가능한 모델로 발전될 수 있을 것이다.
Probiotic의 특성을 뒷받침하는 분자 메커니즘의 여러 가지 측면들이 밝혀지고 있는 실정이다. 기존의 연구들은 단일 유전자와 단백질의 분자 분석에 의존하는 형식이었다. 하지만 지난 10년 전부터 새롭게 선보인 omics 기술의 출현으로 probiotic 미생물의 연구가 전체 게놈 수준에서 가능하게 되었다(Gueimonde and Collado, 2012). 오늘날, metagenomics 방법은 복잡한 생태계 및 그들의 생물의 조성을 밝히는데 또한 사용되고 있다(Qin et al., 2010; Upadrasta et al., 2011). 인체의 모든 부분들은 특정 구획, 연령과 개인의 식습관 그리고 기타 여러 요인에 따라서 다양하게 나누어진 미생물군집을 가지게 된다(Ravel et al., 2014).
미생물 배양액이 순수하거나 또는 적은 양이 포함된 조건들을 시뮬레이션한 것과 비교하여서 Meta-Omics는 probiotics의 in vivo 작용을 이해하기 위한 가장 적절한 첫 번째 방법이다. Omics 기술은 미생물의 유전체 서열보다 저렴하기에 때문에 일반적인 방법으로 널리 이용되어지고 있다(Papadimitriou et al., 2015). 데이터베이스에 대용량처리 transcriptomics 및 proteomics을 통해서 새로운 기능 데이터의 통합은 궁극적으로 후보 미생물들의 probiotic으로서의 잠재력을 컴퓨터 시뮬레이션과 같은 가상 환경에서의(in silico) 평가를 용이하게 할 수 있을 것이다. 하지만, in silico 분석만으로 실험적인 방법을 대체할 수 있다는 전망은 아직은 너무 성급한 예측으로 사료된다. 왜냐하면 probiotic 성질의 분장생물학적 이해가 증가될수록 더 효율적이고 더 정교하게 설계된 in vivo와 in vitro 검사를 설계할 수 있기 때문이다(Papadimitriou et al., 2015). 마지막으로, 인간 미생물군집의 목록표 만드는 작업은 이미 잘 알려진 유산균과 bifidobacteria뿐만 아니라, probiotic으로서의 잠재력을 가진 미생물들까지 새로운 목록으로 고려되어서 연구가 진행되어야 할 것이다.
결 론
본 총설 논문에서는 새로운 probiotic 균주의 발견과 검출을 위해서 현재 사용되고 있거나, 앞으로 사용될 수 있는 다양한 방법들을 자세하게 소개하였다. 물론 여기에 적용할 수 있는 검사방법은 매우 다양하며 많은 것은 사실이지만, 이것들의 능력 면에서는 많은 차이를 보인다(Papadimitriou et al., 2015). 어떤 일부의 분석방법들은 균주의 수가 매우 많을 때 매우 적합한 검사방법이며, 반대로 어떤 분석방법들은 균수가 극히 적은 probiotic 성질을 검증하는데 더 적합하기도 하다. 후보 균주들을 직접 검사하는 방법 외에는 probiotics을 선택하기 위한 완벽한 절차나 순서는 없다(Rijkers et al., 2010). 그러나, 인체 시험의 한계를 고려한다면, 새로운 omics 접근방식과 함께 in vitro와 in vivo는 중요한 부분이 될 것이다. 인간의 건강을 위해 probiotics를 최대한 활용하기 위해서는 방법론적인 진보(발전)이 반드시 필요하며 진행되어야 하는 부분이다. 예를 들면, 새로운 인간화된 동물 모델은 숙주-미생물간의 상호작용을 연구하는데 반드시 필요할 것이다. 만약 probiotic 연구에서 기술적이고 과학적인 해결을 충족할 수 있기 위해서는, 이러한 기술적인 발전은 반드시 관련 법규와 윤리문제 등을 함께 해결하도록 상호 협력하여 진행되어야 할 것이다(Papadimitriou et al., 2015).
오늘날 대부분의 서구 국가에서는 건강 기능성 강조표시(health claim)를 전부 인정하는 것은 현실적으로 매우 어렵다. 특히 유럽에서는 소비자들에게 광고할 때 “probiotic”이란 용어를 사용할 수 없도록 법으로 금지하고 있다. 식품영역에 있어서도 요구르트의 유당불내증에 대한 건강 기능성 강조표시(health claim)를 제외하고 300개 이상의 건강 기능성 강조표시(health claim)를 전혀 승인을 받지 못한 상태이다(Papadimitriou et al., 2015). 숙주와 미생물군집과의 더 자세한 반응 메커니즘과 더 심도 깊은 기능과 역학관계를 이해한 후에 관련 필요한 자료 등을 건강 기능성 강조표시(health claim) 평가 위원들에게 제공하였을 때 건강 기능성 강조표시(health claim)를 법적으로 승인받는데 문제가 없을 것으로 기대된다. 특히, 본 총설논문에서 언급된 다양한 기술적인 진보(발전) 등이 향후 이런 것들을 담당하는데 크게 기여할 것으로 사료된다. 현재 상업적으로 아직 이용되고 있지 않은 F. prausnitzii 또는 A. muciniphila와 같은 것들이 새로운 형태의 probiotics로 고려되어지는 것은 매우 중요하다(Papadimitriou et al., 2015).
본 총설논문에서는 생산공정의 중요성에 관해서는 거의 다루어지지 않았다. 향후에 극도의 혐기적 미생물들인 F. prausnitzii 또는 A. muciniphila을 고려한다면, 소비자 또는 환자들에게 다양한 활력 균주의 전달을 확신할 수 있는 새로운 생산공정, 개량된 저장기술 그리고 관리 전략 등을 충족시킬 수 있는 probiotic 생산은 매우 중요한 부분이 될 것이다. 여러 논문들에서도 probiotic을 준비할 때 산업적 처리공정은 숙주의 기능성에 근본적인 영향을 준다는 것들이 이미 밝혀졌다(Lebeer et al., 2011; 2012; Bron et al., 2012). 왜냐하면 생존 능력, 섬모의 유무, 세포벽 조건, probiotic의 매트릭스 또는 성장단계 등의 여러 가지 요인들은 숙주와 상호작용과 역할에 중요한 영향을 미칠 수 있기 때문이다. 따라서 probiotic의 작용 메커니즘이라 함은 생산공정에서 결정적인 매개 변수들을 포함하고 있음을 의미하는 것이다(Papadimitriou et al., 2015).
의심할 여지도 없이, omics 기술의 지속적인 개발은 전통적인 in vitro와 in vivo 모델이 현재 직면하고 있는 한계성을 완화하는데 도움을 줄 수 있을 것이다. 비록 게놈 및 metagenomic 정보로부터 직접 probiotic 기능성을 예측할 수 있기 전에는 시간이 많이 소요될 수도 있겠지만, 관심 있는 in vitro 또는 in vivo 연구에서 omics 방식의 연속 사용은 향후 probiotics 분야에서 연구 진행속도를 급속하게 향상시킬 수 있을 것으로 사료된다.
요 약
지난 수십 년 동안 행복과 건강에 있어서 식품의 긍정적인 역할에 대한 소비자들의 관심과 인식의 증가 등의 이유로 기능성 식품의 생산 방향으로 식품산업이 변화되어가고 있다. Probiotic 식품의 정의에 의하면, 소비자들의 건강에 도움을 줄 수 있는 충분한 양의 살아있는 미생물을 반드시 포함해야 된다고 규정하고 있다. 오늘날 많은 probiotic 식품들이 판매되고 있으며, 또한 다양한 probiotic 균주들은 상업적으로 이용되고 있다. 하지만, 미생물들의 실제적으로 잠재적인 능력을 어떻게 평가하는 것은 매우 관심을 가지는 부분이다. 왜냐하면 최근 관련 문헌의 검사에서도 알 수 있듯이, probiotic 관련 연구가 급속하게 증가하고 있기에 더욱더 이 부분은 중요하게 인식되어지고 있다. 비록 대부분의 probiotic 미생물들은 식품 또는 공생세균으로서 일반적으로 안전하다고 여겨지고 있지만, 그 외의 재료들에서 얻은 probiotics는 법적인 규제와 안전문제에 대한 우려가 더욱더 증가되고 있는 것은 사실이다. Probiotic으로서 잠재력을 가진 균주들은 in vitro 또는 in vivo 검사를 통해서 선별되어질 수 있다. 예를 들면, 위장 또는 담즙과 같은 산성의 조건에서도 생존능력은 간단한 실험을 통해서 평가될 수 있으며, 또는 면역 활성, 신진대사 기능 또는 장-뇌 상호작용과 같은 복잡한 숙주 기능에서도 영향력의 평가가 가능하게 이루어질 수 있다. 인간의 건강 증진을 위해서는 반드시 고려되어야 하는 것은 궁극적으로 인간을 대상으로 진행되는 임상시험이지만, 지금까지 긍정적인 결과를 나타내는 연구를 통해서 밝혀진 소수의 균주들만이 법적으로 건강 기능성 강조표시(health claim)를 획득할 수 있었다. 따라서 현재 probiotics라고 규정하는데 이용되는 검사방법들의 유효성에 대한 관심이 증가하고 있는 것이 사실이다. 따라서 본 총설논문에서 probiotics의 선별에 이용되는 가장 일반적인 방법과 이들 방법들의 장점 및 한계성에 관해서 자세하게 설명하였다. 더 나아가서, 최근에 omics 기술의 출현은 probiotics의 생물현상을 새롭게 이해하는데 큰 도움으로 주고 있으며, 결국 omics 기술은 probiotics와 같은 다양한 미생물들을 연구하고 선별하는데 새로운 방법으로 이용될 수 있을 것이다. 하지만 여기에 대한 추가적인 연구들은 반드시 진행되어야 할 것이다.






