서 론
통계에 따르면 2015년 330건의 식중독 사건과 이로 인한 5,947명의 식중독 환자가 발생한 것으로 집계된다(Ministry of Food and Drug Safety, 2016). 식중독 발생에 의한 사회경제적 손실은 연간 약 9천 5백억 원에 다다르며(Shin et al., 2010), 식중독균 환자 중 80%가 식중독균 감염 및 중독에 의해 발생한 것으로 보고되었다(Ministry of Food and Drug Safety, 2016). 식중독균에 의한 식중독 발생을 예방하기 위한 정부 및 식품업계 차원의 노력이 투자되고 있다. 식중독에 의한 식중독 발생은 오염식품 검출과 오염식품의 소비 및 섭취 전 배제에 의해 예방된다. 과거 식품 내 식중독균 검출은 배지배양법에 의존하여 수행되었으나, 현재 분자생물학적 기법 기반의 PCR 및 real-time PCR이 병행되고 있으며, 그 결과 식중독균 분석의 속도 및 민감도의 발전이 이루어졌다(Rasmussen et al., 1994; Lazcka et al., 2007; Johnson et al., 2013).
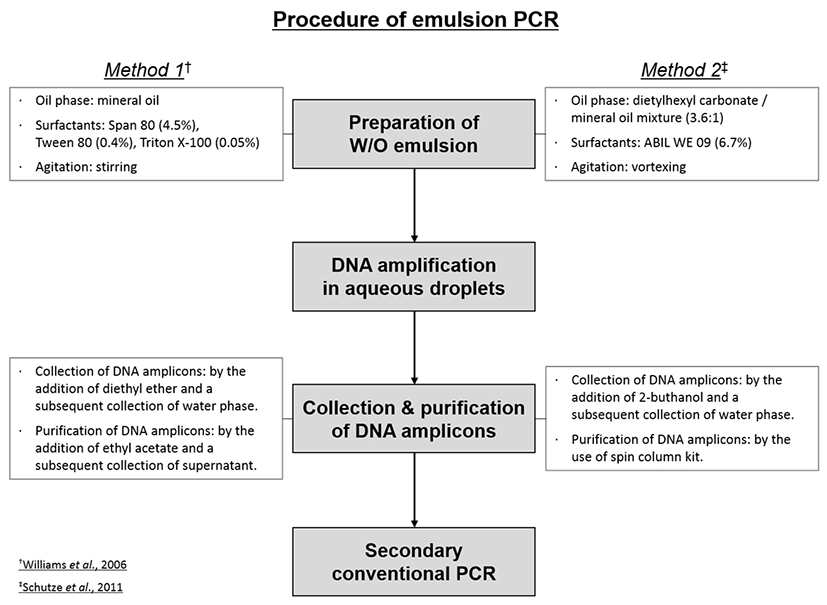
PCR 기법을 이용한 식품 내 식중독균 검출은 식중독균 DNA와 DNA 서열에 상보적인 target-specific primers 및 DNA polymerase의 상호작용에 따른 식중독균 DNA 증폭에 의해 수행된다. Target-specific primers를 이용한 PCR 반응은 대단히 민감하여 1개 혹은 수개의 target DNA를 원활하게 증폭시킬 수 있다. 그러나 PCR 반응은 다양한 유기물․무기물에 의해 간섭을 받을 수 있으며, non-target DNA가 다수 존재할 경우 PCR specificity 및 sensitivity가 현저히 저하된다(Fig. 1). DNA 추출 과정 중 식품성분뿐만 아니라, 식품 내 동식물 조직 및 미생물 DNA가 DNA 시료에 불가피하게 혼입된다. 실질적으로 PCR을 이용한 식품 내 식중독균 분석 중 식품 구성성분과 non-target DNA의 PCR 간섭에 따라 식중독균 DNA가 증폭되지 않는 현상(위음성; false-negative)이 빈번하게 발생하며, 식중독균 위음성 발생은 공공위생 및 식품안전의 커다란 위험요소이다.
우유에 존재하는 plasmin, lactoferrin 및 IgG(Rådström et al., 2004; Powell et al., 1994)과 식육 및 해산물에 존재하는 glycogen 및 탄수화물(Abolmaaty et al., 2007), 채소 및 과일에 존재하는 phonoles, pectin(John, 1992; Sipahioglu et al., 2006)은 대표적인 PCR inhibitors로 알려져 있다. 이와 같은 식품유래 PCR inhibitors는 silica-based column을 이용한 DNA 정제를 통해 상당부분 배제시킬 수 있으나, 식중독균 DNA와 물리․화학적으로 동일한 특성을 나타내는 동․식물 및 non-target 미생물 DNA는 배제는 원천적으로 불가능하다. 특히 식중독균 DNA 개수에 비해 많은 수의 non-target DNA가 PCR 반응에 포함될 경우, 식중독균 DNA가 증폭되지 못하는 결과를 초래한다(Fig. 1)(Meyerhans et al., 1990; Nakano et al., 2003; Chai and Oh, 2015).
Target DNA, primers 및 DNA polymerase 등 PCR 구성요소는 나노단위로 존재한다. 따라서 PCR 반응공간을 물리적 방법을 이용하여 마이크로 단위로 분획하여 DNA를 증폭할 경우 독립적인 target DNA 증폭이 가능하고, PCR inhibitors 및 non-target DNA의 PCR 간섭을 최소화 할 수 있다. PCR 반응공간 및 target DNA는 microwell array 또는 water-in-oil (W/O) emulsion을 이용하여 분획될 수 있으며(Nakano et al., 2003; Hori et al., 2007; Wiktor et al., 2015), 두 기법 모두 PCR specificity 및 sensitivity 향상에 기여하는 것으로 알려져 있다(Margulies et al., 2005; Shendure et al., 2005; Shao et al., 2011; Wiktor et al., 2015). 특히 emulsion PCR(ePCR)로 일컫어지는 W/O emulsion을 이용한 PCR 반응공간 및 target DNA 분획과 PCR 증폭기법은 추가적인 장비구축 없이 기존 PCR 장비를 이용하여 수행될 수 있음에 따라 확장성이 우수하다.
본 논문은 ePCR 기법을 소개하고, ePCR의 식중독균 검출기법 적용에서 고려될 세부요소를 논의함과 동시에 식품안전분야에서 ePCR 기법의 향후 발전가능성을 전망하고자 한다.
Emulsion PCR 적용에 따른 PCR 능력향상의 이론적 배경
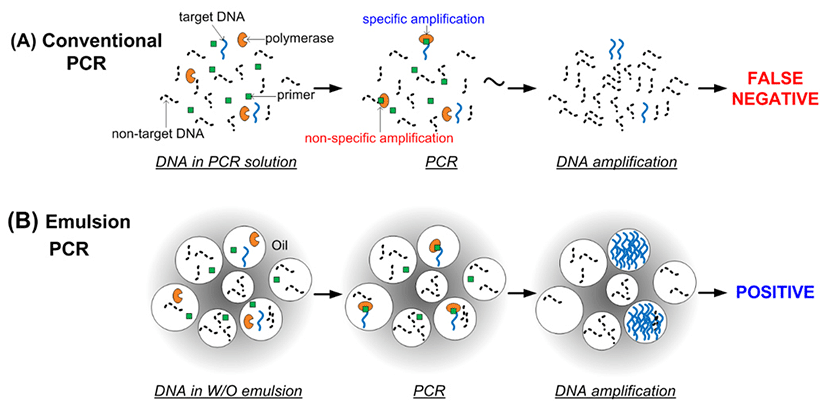
물, oil, 그리고 유화제를 혼합하여 교반하면 작은 크기의 물 droplets을 oil 상에 분산시킬 수 있다. W/O emulsion 내 물 droplets의 수와 크기는 물, oil, 유화제의 비율 및 유화제의 종류에 의해 조절될 수 있으며(Singh and Heldman, 2013), 특정 조건에서 마이크로 단위를 나타내는 약 1010개 수준의 물 droplets을 oil 상에 분산시킬 수 있다(Swami et al., 2008). DNA, primers, DNA polymerase, dNTPs와 같은 PCR 요소는 수용성을 나타내고, 물, oil 및 유화제와 함께 교반할 경우 water phase로 이행한다. 따라서 PCR 구성물을 W/O emulsion에 적용하면 수용성 PCR 요소를 W/O emulsion에 존재하는 물 droplets에 제한하여 분산시킬 수 있다. 일반적으로 PCR solution은 PCR 반응 당 약 4×1010개의 DNA polymerase와 약 2×1017개의 primers, 그리고 약 1×1020개의 dNTPs가 포함된다. 반면, 식품에서 추출된 DNA 개수는 mL당 1010개를 넘지 않고, 20 또는 25 μL의 PCR solution에 약 1~2 μL 수준으로 첨가됨에 따라 식품에서 추출된 DNA는 PCR 반응 당 108개 혹은 미만이 첨가된다. 결과적으로 식품에서 추출된 DNA와 PCR solution을 약 1010개의 물 droplets를 포함하는 W/O emulsion에 분산시킬 경우, 모든 물 droplets은 DNA polymerase, primers 및 dNTPs를 함유하는 반면, DNA는 물 droplets 중 일부에 포함된다. 이 경우 DNA를 포함하는 물 droplets의 개수는 DNA 농도에 의존적이다. W/O emulsion 내 물 droplets의 크기가 일정하고, DNA의 개수가 물 droplets의 개수에 비해 적을 경우, DNA를 포함하는 물 droplets 개수는 Poisson distribution을 따르며, DNA가 k개 존재하는 물 droplets 개수의 percentage는 Poisson equation(Equation 1)에 의해 산출될 수 있다(Swami et al., 2008).
λ: W/O emulsion 내 물 droplets 개수와 총 DNA 개수의 비
e: 자연상수 (~2.718)
W/O emulsion 내 물 droplets의 수가 1010일 때 k개의 DNA를 함유하는 물 droplets 개수에 대한 percentage를 Equation 1을 이용하여 산출하였고, 그 결과를 Fig. 2에 도시하였다. k의 증가에 따라 물 droplets 개수의 percentage는 10진 배수 이상으로 감소하는 것으로 산출된다(Fig. 2). 만약 총 107개의 DNA를 1010개의 물 droplet에 분산시킬 경우, 1개의 DNA를 함유하는 물 droplets는 총 droplets 개수의 약 0.1%를 차지하는 반면, 2개의 DNA를 함유하는 droplets은 5×10-5%를 차지하고, 3개의 DNA를 함유하는 droplets은 약 1.7×10-8%를 차지하는 것으로 산출된다(Fig. 2). 따라서 1개의 물 droplet이 1개의 DNA를 함유할 가능성은 2개 혹은 그 이상의 DNA를 함유할 가능성에 비해 약 2,000배 이상 높다. 결과적으로 DNA와 PCR solution을 W/O emulsion 상에 유화시킬 경우, 첨가된 대부분의 DNA가 개별적으로 물 droplets에 분획될 수 있다(Swami et al., 2008). DNA가 개별적으로 물 droplets에 분획될 경우, non-target DNA 및 PCR inhibitors의 PCR 간섭을 최소화할 수 있으며, target DNA의 독립적 PCR 반응을 유도할 수 있다 (Fig. 1)(Nakano et al., 2003; Hori et al., 2007).
Emulsion PCR 설계 및 수행
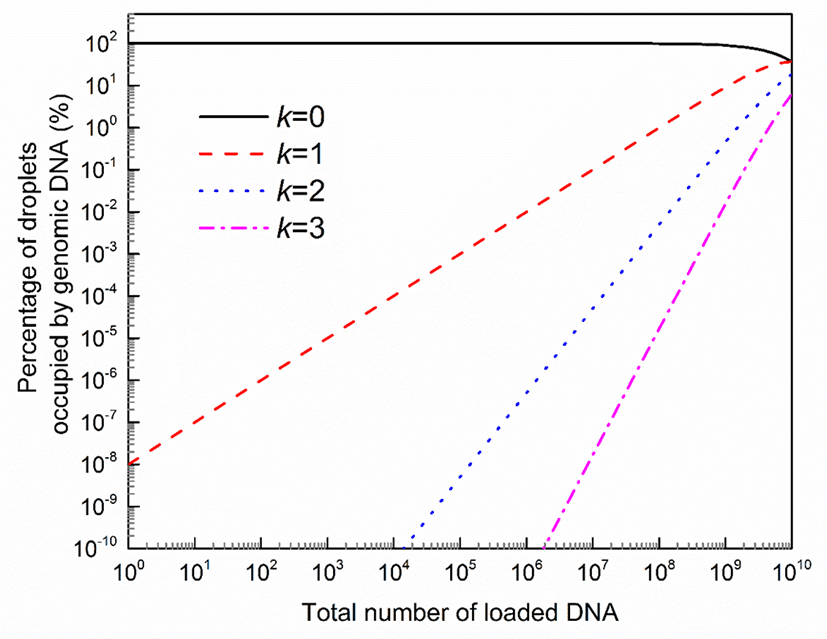
ePCR 과정은 1) oil phase 내 PCR solution 분산을 위한 W/O emulsion 제조, 2) W/O emulsion에 분산․분획된 target PCR의 PCR 증폭, 3) W/O emulsion 내 PCR amplicons 회수 및 정제, 그리고 4) 2차 conventional PCR로 구성된다(Fig. 3).
Target DNA와 PCR 요소의 W/O emulsion 분산 및 물 droplets 내 분획에 따라 PCR 능력 즉 PCR specificity 및 sensitivity의 향상을 기대할 수 있다 (Fig. 1)(Margulies et al., 2005; Shendure et al., 2005; Shao et al., 2011). 그러나 PCR 증폭 공간은 물 droplets 내부공간에 제한되어 최대 PCR amplicons 개수에 대한 제약이 따른다. W/O emulsion 상의 물 droplets의 크기는 마이크로 단위로써(Hori et al., 2007), W/O emulsion에 분획된 target DNA을 15~20 thermal cycles에 의해 PCR 증폭을 유도하면, 물 droplets 내부공간이 PCR amplicons에 의해 가득 채워지고, 더불어 20 thermal cycles 이상의 PCR 수행은 non- specific PCR 반응빈도를 높이는 것으로 알려져 있다(Williams et al., 2006; Schutze et al., 2011). 따라서 W/O emulsion에 분산된 target DNA의 PCR 증폭을 위한 thermal cycles는 20 또는 그 이하 수준으로 적용된다(Williams et al., 2006; Schutze et al., 2011). 15~20 PCR thermal cycles에 의해 생산된 PCR amplicons 개수는 전기영동에 의해 증폭을 확인할 수 있을 수준에 미치지 못함에 따라 추가적인 PCR 증폭과정(2차 conventional PCR)이 요구된다. 2차 conventional PCR 이전 W/O emulsion 상의 PCR amplicons를 수집․정제를 위한 과정이 필수적이며, 유기용매(diethyl ether 또는 2-buthanol)를 이용한 water phase 수집과 water phase 내 PCR amplicons 정제를 위한 DNA purification 단계가 본 과정에 포함된다(Fig. 3). W/O emulsion 상의 target DNA 증폭을 위한 PCR과 2차 conventional PCR에 적용되는 thermal cycling 조건은 일반적 PCR 조건과 유사하다. 그러나 W/O emulsion 상의 물 droplet에 분획된 target DNA의 PCR 증폭과정 중 W/O emulsion의 안정성 유지를 위해 DNA denaturation 단계의 온도를 95℃ 이하로 설정하는 것이 보편적이다(Williams et al., 2006; Schutze et al., 2011).
Target DNA는 PCR solution 및 oil phase의 혼합․교반에 따라 W/O emulsion에 분산되고, 마이크로 단위의 물 droplet에 분획된다. Water phase인 PCR solution을 물리․화학적으로 상이한 oil phase에 분산하기 위해 양친매성의 유화제가 사용된다. 유화제는 water-oil 계면상에 존재하며, 친수성 head와 소수성 tail 부분이 각각 water phase 및 oil phase로 이행하여 water phases와 oil phase간의 표면장력의 차이를 감소시키고, water phase의 oil phase 내 분산을 유도한다. 유화제를 이용하여 target DNA 및 PCR 요소를 oil phase 내부에 형성되는 물 droplets에 분획할 때, 물 droplets 계면상에서 water phase로 이행된 유화제 head는 target DNA 및 PCR 요소와 직접적으로 접촉할 수 있고, 물리․화학적으로 반응할 수 있다. 물 droplets에 분획된 target DNA의 PCR 반응 중 유화제 head에 의한 의도치 않은 반응을 최소화하기 위해 화학적으로 반응성이 적은 비극성 유화제를 이용한 W/O emulsion 제조가 바람직하다. 보고에 따르면 span 80, tween 80, triton X-100과 같은 비극성 유화제 및 ABIL EM 90, ABIL WE 09와 같은 비극성 유화제 복합물이 ePCR에 주로 적용된다(Williams et al., 2006; Schutze et al., 2011; Murgha et al., 2014). ePCR에 적용되는 유화제의 농도는 oil phase 대비 최대 10% v/v를 넘지 않으며, mineral oil을 oil phase로 적용할 경우 mineral oil 5% v/v 이하(Williams et al., 2006), 그리고 dietylhexyl carbonate /mineral oil 혼합물을 oil phase로 이용될 경우 7% v/v 수준으로 유화제가 첨가된다 (Fig. 3)(Schutze et al., 2011).
ePCR에 적용되는 oil phase의 경우, 화학적 안정성이 우수한 mineral oil이 보편적으로 사용되나, 실험적 편의성 향상을 위해 흐름성과 안정성이 우수한 dietylhexyl carbonate을 mineral oil과 혼합하여 사용되기도 한다(Williams et al., 2006; Schutze et al., 2011). 또한 PCR 수행 간 W/O emulsion 안정성은 water phase와 oil phase 비율에 의해 결정되며, mineral oil을 oil phase로 적용할 경우 1:2(water phase:oil phase)(Williams et al., 2006), 그리고 dietylhexyl carbonate / mineral oil 혼합물을 oil phase로 이용될 경우 1:6 수준의 water phase(PCR solution) 첨가가 권장된다(Schutze et al., 2011; Chai and Oh, 2015). 1:2 또는 1:6 수준 이상으로 water phase를 첨가할 경우, water phase의 상분리(phase separation) 현상이 관찰된 바 있다(unpublished data).
W/O emulsion 상의 물 droplets에 분산․분획된 target DNA 및 PCR 요소는 water-oil 계면으로 확산될 수 있다. 또한 target DNA 및 PCR 요소와 oil phase의 직접적 접촉은 PCR 반응에서 부정적 결과를 초래한다. 물 droplets에 분산․분획된 target DNA 및 PCR 요소와 oil phase의 직접적 접촉은 bovine serum albumin(BSA)에 의해 방지될 수 있다(Williams et al., 2006; Schutze et al., 2011). 따라서 ePCR 설계에서 BSA와 같이 target DNA 및 PCR 요소와 oil phase의 직접적 접촉을 방지할 수 있는 물질 첨가를 고려해야 한다.
W/O emulsion에 의한 target DNA 및 PCR 요소 분산․분획은 PCR specificity 및 sensitivity 향상을 유기하지만(Margulies et al., 2005; Shendure et al., 2005; Shao et al., 2011), 앞서 언급한 바와 같이 PCR 반응공간이 마이크로 단위의 물 droplets에 제한되어 최대 PCR amplicons 개수에 대한 제약이 따른다(Williams et al., 2006; Schutze et al., 2011). 바꾸어 기술하면 물 droplets 내 PCR 반응공간은 primers, DNA polymerase, dNTPs 및 BSA의 농도에 영향을 받는다. 특히 물 droplets 내 target DNA 증폭 효율은 primers와 dNTPs에 비해 부피가 큰 DNA polymerase와 BSA의 농도에 민감하게 영향을 받는 것으로 알려져 있다(Schutze et al., 2011; Shao et al., 2011). W/O emulsion 상의 물 droplets에 분산․분획된 target DNA 증폭 적용을 위한 DNA polymerase의 농도는 conventional PCR에 비해 다소 낮은 0.6~1.5 U(PCR 반응 당) 수준이 적용되며(Schutze et al., 2011), BSA 농도는 PCR solution μL 당 5 μg 수준으로 첨가된다(Schutze et al., 2011). 그러나 각 시료조건에 따라 ePCR을 위한 적정 DNA polymerase 및 BSA 농도는 실험을 통해 도출하는 것이 바람직하다. 참고적으로 ABIL WE 09를 이용하여 dietylhexyl carbonate/mineral oil 상의 물 droplets에 분산․분획된 E. coli O157:H7 DNA는 BSA 0.48 μg/μL 및 Taq DNA polymerase 1.25 U 조건에서 원활히 증폭된 바 있다(Chai and Oh, 2015).
Emulsion PCR의 적용과 전망
W/O emulsion에 의한 DNA 분획에 따른 target DNA의 독립적 PCR 반응결과, 고도의 PCR specificity와 sensitivity를 기대할 수 있다(Margulies et al., 2005; Shendure et al., 2005; Shao et al., 2011). Conventional PCR 과정 중 빈번하게 발생하는 non-specific PCR 반응은 다수의 non-target DNA에 혼합된 10개 미만의 target DNA 증폭을 방해하여 target DNA 위음성 발생 빈도를 증가시키는 반면, W/O emulsion에 의해 target DNA를 마이크로 단위의 물 droplets에 분획할 경우, 다수의 non-target DNA에 혼합된 1개 또는 10개 미만의 target DNA를 선별적으로 증폭할 수 있다(Shao et al., 2011; Chai and Oh, 2015). ePCR 적용에 의한 PCR specificity 및 sensitivity 향상은 향후 식품 미생물 안전 진단의 민감도 향상과 위음성 발생 빈도 저하에 기여할 것으로 판단된다.
ePCR은 aptamer libraries 제작에 유용하게 사용될 수 있다 (Shao et al., 2011). Aptamers는 바이오센서 등에 사용되는 RNA 또는 DNA 절편으로써 무작위적 서열과 특정 서열이 혼합된 다수의 template DNA 합성(DNA libraries 제작)과 합성된 template DNA 증폭에 의해 생산된다. Conventional PCR을 이용하여 상이한 서열을 포함하는 다수의 합성 template DNA를 증폭할 경우, 초기 특정 template DNA에서 증폭된 aptamers이 반복적으로 증폭되어 특정 aptamers 우점 현상이 발생한다. 만약 목적으로 하는 서열의 aptamers가 PCR amplicons에서 발견되지 않을 경우, 목적 aptamers 생산을 위해 DNA libraries 제작 및 conventional PCR을 반복적으로 수행해야 하는 등 많은 어려움이 따른다. 그러나 ePCR을 적용할 경우, W/O emulsion에 분획된 template DNA 증폭이 독립적으로 진행되어 특정 서열의 aptamers 우점 발생을 방지하므로 풍부한 aptamers를 포함하는 libraries 작성이 가능하다. 따라서 ePCR을 DNA libraries에 적용할 경우, aptamers 개발의 시간 및 인력 절감효과를 꾀할 수 있다(Shao et al., 2011).
또한 ePCR은 microbiome 분석을 위한 pyrosequencing과 meta 분석을 가능하게 하는 핵심 수단으로 이용되고 있다(Schuster, 2008). 환경 또는 생체 내 microbiome은 다양한 미생물에 의해 복잡하게 구성되어 있다. Microbiome의 pyrosequencing 및 meta 분석은 microbiome으로부터 추출된 미생물 DNA의 서열분석에 의해 수행된다. 그러나 정확한 서열분석을 위해 적정 수 이상의 DNA templates를 필요로 한다. 환경 또는 생체 시료로부터 추출된 미생물 DNA의 개수를 enrichment하기 위하여 conventional PCR을 적용할 경우, 우점 미생물 DNA가 반복적으로 증폭되어 작은 군집을 가지는 미생물 DNA의 sequencing이 무시되는 결과를 나타낸다. ePCR은 적은 수의 DNA를 효율적으로 증폭할 수 있음에 따라 pyrosequencing 중 DNA enrichment에 빈번하게 응용․적용되고 있다(Matzas et al., 2010; Siqueira et al., 2012). 비록 pyrosequencing에 적용되는 ePCR은 앞서 기술된 기법과 세부적으로 차이를 나타내나, 이론적, 기술적으로 동일한 배경에 의해 수행된다.
최근 SYBR-GREEN과 같은 형광물질을 ePCR에 적용하여 물 droplets 내부의 DNA 증폭 유무를 직접 확인할 수 있는 기법에 대한 연구가 활발히 진행되고 있다(Schuler et al., 2016). 형광물질을 이용한 물 droplets 내 DNA 증폭 확인기법은 W/O emulsion 내 PCR amplicons 회수 및 정제 그리고 2차 conventional PCR 단계를 필요로 하지 않기 때문에 당 기법의 개발에 따라 분석비용, 분석시간, 인력 절감을 꾀할 수 있다. 또한 DNA가 증폭된 물 droplets 개수를 계수하면 ePCR 시료 내 target DNA 개수를 산출할 수 있다(Miotke et al., 2014; Corbisier et al., 2015). 비록 아직까지 보고된 바 없으나, 당 기법은 식품 내 식중독균 정량기법에 적용될 수 있을 것으로 기대되어 식품 미생물 안전성 향상에 도움이 될 것으로 전망된다.