서 론
Cryptic prophage는 파지의 구조 유전자 결실이나 핵심 조절 유전자의 비활성화로 인해 더 이상 감염성 파지를 생성하지 못하면서, 숙주 유전체에 안정적으로 삽입되어 존재하는 파지 DNA이다[1]. 반면 prophage는 비활성화된 상태로 숙주 유전체에 존재하지만, 특정 자극하에서 유도되어 용균주기(lytic cycle)로 전환하여 새로운 파지 입자를 형성한다[1]. 비활성 상태의 cryptic prophage는 기능하지 않는 ‘genomic junk’로 간주되어 상대적으로 주목받지 못하였으나[2], 최근에는 숙주의 유전자 발현 조절, 스트레스 반응, 항생제 저항성, 바이오 필름 형성 등 다양한 생물학적 과정에 관여할 수 있음이 보고되고 있다[3]. 관련 연구는 주로 Escherichia coli에서 활발히 수행되어 왔으며[3–7] 현재는 cryptic prophage가 프로바이오틱스와 같은 산업적으로 유용한 균주에서도 면역 관련 유전자 발현 유도[8], 스트레스 방어 기작 유도[9], ‘superinfection’ 감염 방지[10] 등의 기능적 역할을 수행할 수 있는 가능성이 제기되고 있다.
유산균(lactic acid bacteria, LAB)은 발효식품 산업과 프로바이오틱스의 주요 균주군으로 장 미생물총 조절을 통한 장 건강 개선, 면역 조절 등의 기능성과 더불어 발효식품의 품질 개선 등 다양한 이점을 제공한다[11,12]. LAB 유전체에는 파지(bacteriophage) 기원 유전자들이 광범위하게 존재하며[3], 그중 상당수가 prophage 형태로 게놈에 통합되어 있다[13]. 실제로 발효유 제품에서 분리한 Lactobacillus rhamnosus 및 Lactobacillusdelbrueckii 균주의 전장유전체 분석(whole genome sequencing, WGS) 결과, 여러 개의 완전형 prophage가 유전체에 통합되어 있는 것이 보고되었으며[14], 895개의 Lactobacillaceae 계통 유전체를 대상으로 한 대규모 in silico 분석에서도 40% 이상의 균주가 prophage를 보유하고 있고, 이 중 61.4%는 실제로 유도 가능한(inducible) 형태로 존재하는 것으로 나타났다[15].
이처럼 LAB 유전체 내 prophage 존재는 매우 보편적인 현상으로 발효 공정에서 발생하는 환경 스트레스로 인한 LAB 유전체 내 prophage의 활성화는 균주의 생산성, 기능, 안정성에 부정적인 영향을 줄 수 있다[16]. 유도된 prophage는 숙주 세포의 용균(lysis)을 유도하여 배양액 내 세포 수를 감소시키며, 이로 인해 젖산 생산성(lactic acid productivity)이 저하되어 발효 지연과 불균일한 제품 생산의 문제가 발생하게 된다[15,16]. 따라서 prophage 존재 여부 및 유도 가능성에 대한 평가는 스타터 균주 선정 시 안정적으로 고품질의 유제품을 생산하는 데 있어 필수적이라 할 수 있다.
한편, 비활성화된 상태의 cryptic prophage는 감염성 입자를 생성하지 않지만, 숙주 유전체 내에 안정적으로 잔존하면서 유전적 재조합과 수평 유전자 이동이 일어나는 유전체적 변이의 중심으로 기능한다는 것이 보고되었다[17,18]. 또한 cryptic prophage가 항생제 내성, 산화·삼투·산성 스트레스 내성과 관련된 유전자를 포함하여, 숙주의 생존과 적응을 매개하는 역할도 수행하는 것으로 보고되었다[17]. 따라서 식품 산업에서 프로바이오틱스 또는 스타터로 사용할 균주를 선택할 때, prophage 및 cryptic prophage 보유 여부가 품질과 식품 안정성 확보를 위한 주요 고려 요소가 될 수 있다.
그럼에도 불구하고 LAB에서 cryptic prophage의 구조적 특성과 기능에 대한 연구는 현재까지도 제한적으로 수행되고 있다. 지금까지의 파지에 대한 연구는 주로 완전한 기능의 prophage 또는 유도 가능한 파지에 초점을 맞춰 왔고, cryptic prophage의 경우는 대부분 분석 대상에서 배제되거나 낮은 비중으로 다뤄졌다[19]. 특히 LAB에서는 유전체 안정성과 숙주의 안전성이 중요함에도, prophage와 cryptic prophage가 숙주에게 미치는 영향을 체계적으로 분석한 사례는 드물다[16].
그중 우리가 주목한 LAB은 Lactiplantibacillus plantarum로 높은 유전체 다양성과 우수한 생존력을 가져 다양한 환경에 적응할 수 있는 대표적인 종이다. 이 중 WCFS1 균주는 2003년에 수행된 Ventura의 연구에서 유전체 내에 4개의 prophage-like 요소를 보유하고 있다고 보고하였다[20]. 해당 연구에서는 ORF(open reading frame) 수준의 비교유전체학(comparative genomics) 접근법을 기반으로, 유전체 내 개별 유전자를 BLAST(basic local alignment search tool) 및 PSI-BLAST(position-specific iterated BLAST)를 통해 분석하여 phage hallmark 유전자의 존재를 확인하였다. 또한 tRNA(Trp, Asn, Ile, Leu, Met, Gly) 서열 및 att(adenine-thymine-thymine) 반복서열 탐색을 통해 prophage의 통합 부위를 추정하였으며, PCR(polymerase chain reaction) bridging을 이용한 삽입 경계 검증과 Northern blot 분석을 통해 전사 활성 여부를 확인함으로써 prophage의 기능적 상태를 평가하였다.
본 연구에서는 WGS이 완료된 L. plantarum WCFS1 균주의 유전체를 대상으로 세 가지 파지 관련 유전자 예측 도구 PHASTEST(phage search tool with enhanced sequence translation)를 활용하여, prophage 존재 여부, 구조 및 기능 유전자의 보존·결손 여부를 종합 평가함으로써 prophage 및 cryptic prophage 요소의 분포와 그로 인한 구조적 특징 그리고 유전자 기능적 특성을 분석하였다. 이는 향후 L. plantarum WCFS1 균주 외에도 LAB의 유전체를 기반으로 한 특성 평가 및 식품 안정성 확보를 위한 새로운 유전적 고려 기준을 제공할 수 있을 것이라 기대한다.
재료 및 방법
L.plantarum WCFS1의 완전 유전체 서열(GenBank accession no. AL935263.2)은 미국 국립생물공학정보센터(NCBI) database에서 제공받았다. 이후 prophage 예측 및 기능 분석을 위해 FASTA(.fna)와 GenBank(.gbk) 형식 파일을 사용하였다.
유전체 내 prophage 영역 예측은 PHASTEST 3.0을 이용하여 수행하였다[21]. 분석 prophage로 예측된 후보 영역은 완전성 점수(0–150)에 따라 “Intact(>90)”, “Questionable(70≤score≤90)”, or “Incomplete(<70)”로 분류하였으며 PHASTEST로 예측된 prophage 영역의 위치와 구조는 원형 및 선형 유전체 지도상에 도식화하였다(Figs. 1 and 2). 예측된 영역 내 유전자 기능 분석은 Prokka 1.14.6[22]와 Bakta v1.8.2[23]를 사용하여 수행되었으며, 프로그램 내장 데이터베이스를 통해 prophage 영역 내 예측 ORF의 유전자명, 단백질 기능을 확인하였으며 기능적 분류를 진행하였다.
결과 및 고찰
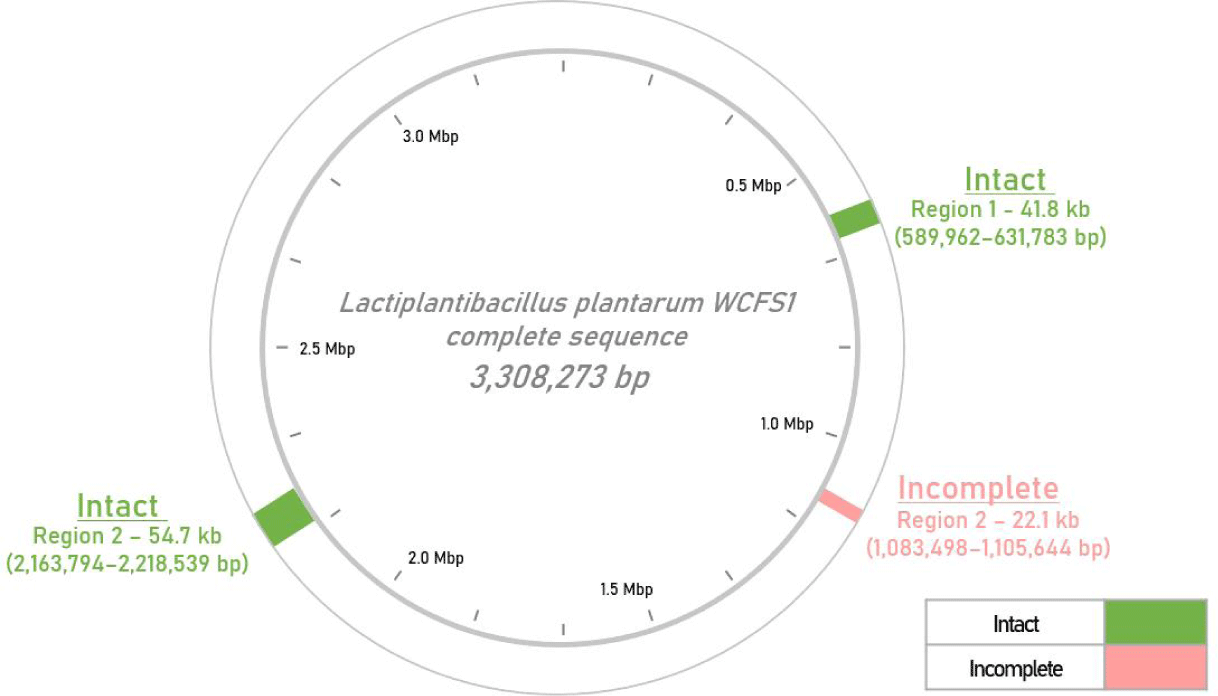
L.plantarum WCFS1의 유전체를 PHASTEST를 이용하여 분석한 결과, 총 3개의 prophage 영역이 예측되었다(Fig. 1). 이 중 Region 1(589,962–631,783 bp; 41.8 kb)과 Region 3(2,163,794–2,218,539 bp; 54.7 kb)은 PHASTEST에서 제공하는 completeness(score) 150점으로 intact prophage로 판정되었으며, Region 2(1,083,498–1,105,644 bp; 22.1 kb)는 50점으로 incomplete prophage로 판정되었다(Table 1). PHASTEST를 통해 예측된 각 영역 내 phage-related ORF 수는 Region 1은 61개, Region 2는 29개, Region 3는 70개로 확인되었다. 2003년에 수행된 Ventura의 연구에서는 L. plantarum에서 총 네 개의 파지 관련 영역(Lp1–Lp4)을 보고하였으며, 해당 영역에서 degraded remnant인 R-Lp4 영역을 제외하고 PHASTEST 분석 결과와 일부 중복되었다[20]. Lp1(약 589–632 kb)는 본 연구의 PHASTEST Region 1, Lp2(약 2.16–2.20 Mb)와 Lp3(약 2.21-2.22 Mb)는 PHASTEST Region 3으로 확인되었다. 한편 Region 2(incomplete prophage) 구간이 PHASTEST를 통해 새롭게 예측되었는데 당시 연구가 PCR bridging이나 Northern blot 기반 분석을 중심으로 유도 가능성(inducibility)과 구조적 완전성(completeness)을 확인하였기 때문에, 불완전한 영역인 Region 2는 검출이 불가능했을 것이라 판단된다.
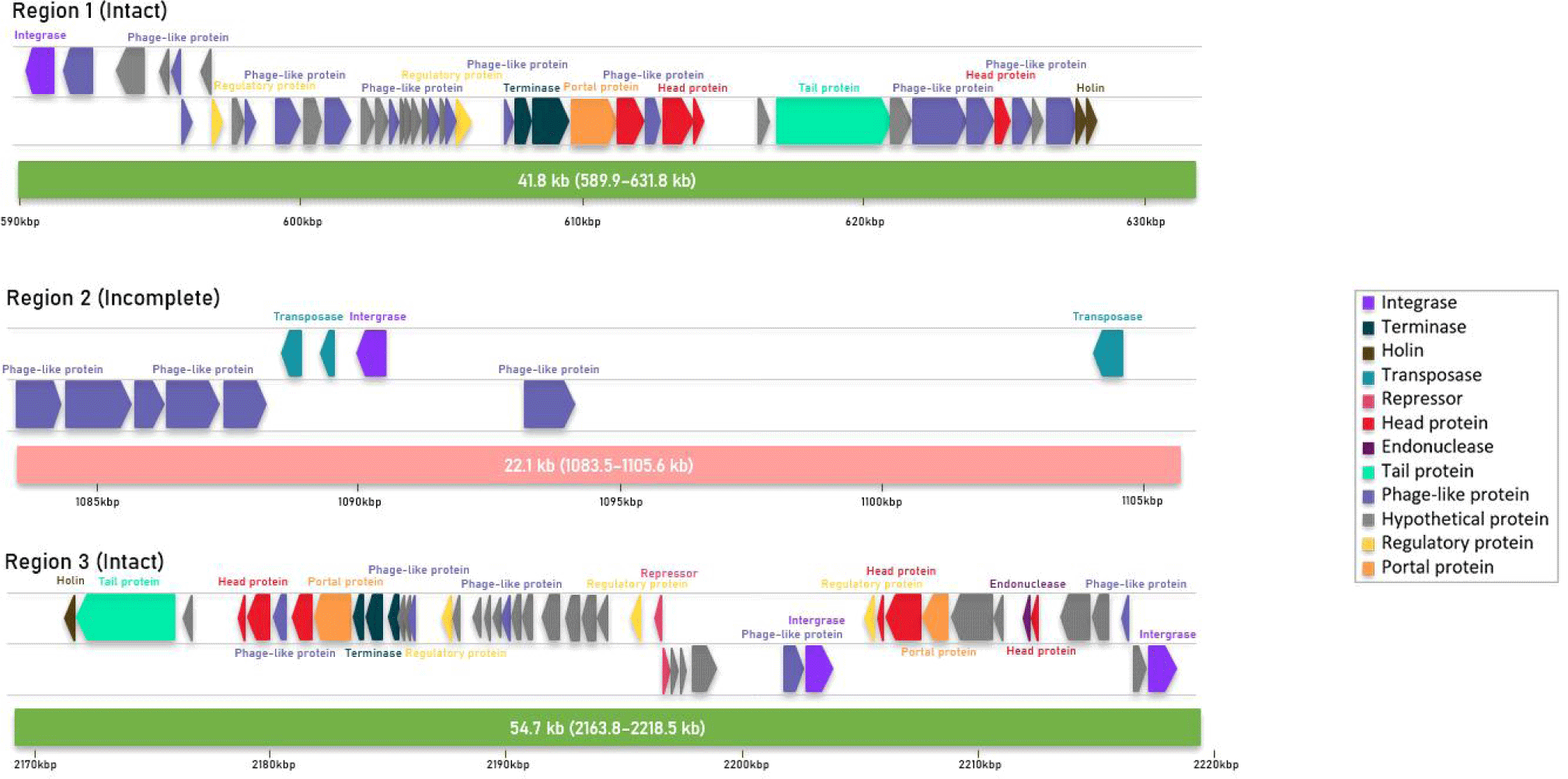
PHASTEST 예측 영역에 대해 Prokka와 Bakta을 통해 유전자 기능을 예측한 결과, PHASTEST Region 1과 3에서는 capsid, tail, portal, terminase 등 파지 입자 형성에 관여하는 구조 유전자(structural genes)와, repressor(CI), Cro, integrase(xerC), antirepressor(arpU) 등 파지의 용원–용균 전환(lysogenic–lytic switch) 생활사를 조절하는 유전자(regulatory genes)가 비교적 완전하게 보존되어 있었다.
반면 Region 2에서는 capsid 및 tail 유전자를 포함한 구조 모듈(structural module)이 결여되어 있었으며, 대신 integrase/recombinase(xerC_3) 및 다수의 glycosyltransferase 계열 유전자 등 대사 및 재조합에 관여하는 유전자(metabolic and recombination-related genes)가 존재하였다(Table 2). 이는 L. plantarum WCFS1 유전체 내에 활성화 가능한 prophage 영역과 비활성화된 cryptic prophage 영역이 공존하고 있음을 시사한다. 한 예시로 발효 대두에서 분리된 L. plantarum 5-2 균주의 comparative genome analysis 연구에서도 세 개의 완전한 prophage와 하나의 불완전한 파지 영역이 발견되었고, 실제로 다양한 Lactobacillus 속 균주에서도 불완전 prophage 영역이 보존되어 있다[24]. 결과적으로 Lactobacillus 속에 불완전 prophage가 보편적으로 존재한다.
Region 1과 Region 3에서 모두 파지 입자 형성에 필요한 핵심 구조 유전자가 온전히 존재하였다(Table 2). 이는 두 영역 모두 lytic induction을 통해 감염성 파지 입자를 형성할 수 있는 온전한 형태의 prophage(intact prophage)임을 의미한다[5,13].
Region 1(589,962–631,783 bp; 41.8 kb)에서는 integrase(xerC), terminase, portal, head, tail, holin 등 파지 입자 형성과 관련된 hallmark 파지 유전자를 포함하고 있다. 또한 lexA/xre, cro/C1-type domain, arpU 등 전사조절 인자(transcriptional regulators)가 함께 존재하여, 해당 영역이 숙주의 lysogenic–lytic 전환 및 lysogenic integration 조절에 관여할 가능성이 있다(Table 3, Fig. 2). 이러한 조절 인자들과 함께, 해당 영역에서는 숙주 상호작용이나 외막 성분 변형과 관련된 보조적 기능 유전자(accessory functional genes)도 함께 확인되었는데 SGNH(serine, glycine, asparagine, histidine)/GDSL(Gly-Asp-Ser-Leu) hydrolase 계열의 단백질이 해당된다. 이들은 구조적으로 탄수화물 변형 효소(superfamily of carbohydrate-modifying enzymes)에 속하고, 세균과 파지 모두에서 다당류 변형(glycan modification)에 관여하는 것으로 알려져 있다[25]. 따라서, 해당 영역에 존재하는 SGNH/GDSL hydrolase 단백질은 외막 다당체 변형을 통한 파지–숙주 상호작용 조절 기능에 관여할 가능성이 있을 것이라 예측된다. 이러한 구성 인자 및 유전자를 고려할 때, Region 1은 파지 입자 형성에 필수적인 구조 유전자를 갖춘 intact prophage로서, 조절 및 보조 유전자의 공존을 통해 파지의 생활사(lysogenic–lytic switch) 전환 조절과 숙주와의 상호작용에 관여할 것으로 예측된다.
Region 3(2,163,794–2,218,539 bp; 54.7 kb)은 Region 1과 동일하게 주요 구조 유전자가 완전하게 보존되어 있다. 이 영역에는 Integrase(xerC_6), terminase large/small, portal, capsid, tail, tape measure, holin, endonuclease 등의 핵심 유전자들과 함께 lexA/xre, arpU 등의 전사 조절 인자(transcriptional regulatory factors), 그리고 다수의 tRNA 유전자가 위치하고 있다(Table 3, Fig. 2). 이와 같이 tRNA 유전자 인접 부위에 integrase 및 조절 인자가 함께 존재하는 배열은 과거 파지 통합에 의해 형성된 전형적인 prophage integration hotspot일 가능성이 높다[13]. 이와 함께 DNA methyltransferase, replication protein, endonuclease 등의 보조 인자(accessory proteins)가 존재하는 것을 보아, 파지 유전체의 안정성 유지, 복제 조절, 및 재조합 활성화에 관여할 가능성이 있는 intact prophage일 것이라 예측된다.
요약하자면, Region 1(589,962–631,783 bp; 41.8 kb)과 Region 3(2,163,794–2,218,539 bp; 54.7 kb)은 모두 L. plantarum WCFS1 게놈 내에서 구조적 완전성(intact structure)을 유지하는 prophage 영역으로 확인되었다. L. plantarum WCFS1은 이전 연구에서 mitomycin C 처리에 의해 해당 영역이 실제로 유도되지 않았으나[20], 이후에 유전적으로 완전한 prophage라 하더라도 mitomycin C에 의해 항상 유도되는 것이 아니라는 것이 증명되었다[26,27]. Lactobacillus reuteri 6475에서 RecA–LexA 의존적 SOS 반응이 phage 유전자 발현을 조절함이 보고된 선행 연구를 고려할 때, L. plantarum WCFS1의 해당 prophage가 LexA/Xre 조절 인자에 의해 잠재적 비활성 상태로 존재할 가능성이 높다[28]. 따라서 Region 1, 3은 mitomycin C 처리에 비유도성이지만, 유전자 구조적 측면에서는 여전히 intact prophage로 분류될 수 있으며, 다른 환경적 스트레스 조건에서 활성화될 수 있는 prophage로 예측한다.
Region 2(1,083,498–1,105,644 bp; 22.1 kb)는 분석결과 구조 유전자가 결여된 incomplete prophage 영역으로 예측되었다. 해당 영역에서는 integrase/recombinase(xerC_3), integrase catalytic domain protein(insK), 그리고 transposase이 존재하고 이는 prophage의 통합 및 재조합 기능을 부분적으로 수행할 것으로 예측되었다(Table 2). 반면 capsid, tail, tape measure, terminase large/small, portal 등 phage particle assembly에 필수적인 hallmark 구조 유전자들이 모두 결여되어 있었으며, holin과 endolysin 또한 존재하지 않아 일반적인 lytic cycle로의 전환이 불가능한 것으로 보인다(Table 3, Fig. 2). 선행 연구들에 따르면 prophage가 capsid, tail, portal, terminase와 같은 필수 구조 모듈을 상실하여 더 이상 유도되거나 phage particles을 생성할 수 없는 경우 이를 기능적으로 ‘cryptic prophage’로 분류한다[1,5,29]. Region 2는 이러한 구조적 결손과 lytic 불능 특성을 모두 충족하므로 incomplete 상태보다는 cryptic prophage로 판단된다. 이러한 구조적 결함에도 불구하고 해당 영역은 다양한 기능성 유전자를 보유하고 있다. rfbA, cpsC, cpsD, cps2G 등의 EPS(exopolysaccharide) 합성 관련 유전자군이 존재하는데, 이는 해당 영역이 세포 표면 다당체 생합성(exopolysaccharide biosynthesis)에 관여하여 장내 정착성 및 환경 적응성 향상에 기여할 것으로 판단된다[28,30]. 이러한 기능성 유전자들의 존재 여부는, prophage 잔존체가 스트레스 내성과 대사 적응성 등의 생리적 조절에 이점을 부여할 뿐만 아니라 유전체 내 변이와 수평적 유전자 이동 촉진을 통한 유전체 가소성(genome plasticity) 증진에 기여할 수 있다는 선행 연구의 보고와 일치한다[18].
해당 영역에는 EPS 관련 유전자 외에도 숙주의 대사 및 신호 조절과 관련된 인자들도 함께 존재하였다(Table 3). 영역 내 확인된 핵심 인자 중 단백질 탈인산화 효소 tyrosine phosphatase(ywqE_1)는 세포 내 단백질 인산화 수준을 조절함으로써, 탄수화물 대사(carbohydrate metabolism), 세포벽 생합성(cell wall biosynthesis), 에너지 생성과 같은 주요 대사 경로의 조절에 관여하는 신호전달 인자로 알려져 있다[31]. 이러한 효소가 prophage 영역 내에서 확인된다는 점은, 해당 유전자가 파지 유래 유전자임에도 숙주의 대사 네트워크 및 생리 조절에 보존적으로 기여할 가능성이 있다. PHASTEST Region 2의 유전자 구성을 종합하여 볼 때 구조적 결함으로 인해 lytic cycle로의 전환은 불가능하나, 숙주 세포 표면 다당체 조절과 대사 네트워크의 신호전달 조절을 매개함으로써, 환경 적응성과 유전체 가소성(genomic plasticity)의 증진에 기여하는 cryptic prophage로 기능할 가능성이 높다.
이러한 구조적·기능적 특징을 확인한 이후, 본 연구에서는 스타터 균주로서의 산업적 안전성을 평가하기 위해 추가적으로 각 prophage 영역에 대한 항생제 내성 유전자(antibiotic resistance genes, ARGs) 및 병원성 인자(virulence factors) 존재 여부를 분석하였다. CARD(comprehensive antibiotic resistance database) Resistance Gene Identifier[32]와 ResFinder[33] 분석 결과, 세 prophage 영역 모두에서 기준 조건에 해당하는 ARG는 검출되지 않았다. VFDB 기반 BLAST 분석[34]에서는 sugar transferase(GBS_RS06590)와 wbaP/rfbP 계열 glycosyltransferase가 낮은 수준의 유사도로 확인되었으나, 이는 병원성과 무관한 일반적인 캡슐·다당체 생합성 관련 대사 유전자임을 확인하였다. 따라서 L. plantarum WCFS1의 prophage 영역에는 식품 스타터 균주의 안전성을 저해할 만한 유전적 요소가 존재하지 않는 것으로 판단된다.
Cryptic prophage는 감염성 입자를 더 이상 형성하지 않지만, 숙주의 생존성과 스트레스 내성, 적응 진화에 관여하는 prophage 잔존체(prophage remnants)로서 기여할 수 있다고 제안되었다[19,35]. Lactobacillus 속 prophage가 숙주 세포의 생리적 과정에 관여한다는 사실이 보고되었다[36]. 해당 연구에서는 Lactobacillus 속 prophage 유전자가 특정 스트레스 조건에서 전사적으로 활성화되는 현상이 관찰되었으며, 이러한 활성은 숙주 세포의 환경 적응을 향상시키고 결과적으로 생존 향상에 기여할 가능성을 제안하였다[36]. 또한 Lactobacillus 속 19종 균주의 유전체 분석을 통해 예측된 prophage 영역에서 총 431개의 ARGs와 virulence 관련 유전자가 확인되어, prophage가 유전자의 수평적 전달(horizontal gene transfer)의 역할을 하는 것이 보고되었다[32]. 해당 결과는 prophage가 영양 결핍, 산화 스트레스 등 생존에 불리한 환경(adverse environments) 속에서 숙주 생존성을 향상시키는 유전적 요소로 작용할 수 있으며, 비활성화된 prophage 잔존체가 숙주의 생리 조절에 기여할 수 있음을 뒷받침한다.
Prophage가 숙주의 생존성과 생리 조절에 기여할 수 있다는 선행 연구들의 결과는, 본 연구에서 L. plantarum WCFS1의 prophage 영역에서 확인된 기능 유전자의 특성과도 일치한다[19,31,32]. L. plantarum WCFS1의 Region 2에서 확인된 integrase는 prophage의 부위특이적 재조합(site-specific recombination)을 매개하는 핵심 효소로서, 숙주 유전체 내 새로운 삽입 또는 재배열을 통해 유전적 다양성과 균주 특이성을 형성하고, 이러한 구조적 변화는 인접 유전자의 발현 또는 기능 변화에 관여할 것으로 예측된다[29,36,37]. 이어서, L. plantarum 유래 prophage에서 mshA 및 cps2G와 같은 EPS 생합성 유전자가 숙주의 생존 이점을 제공할 수 있다고 보고된 연구 결과[36]를 바탕으로, 본 연구에서 확인된 EPS 생합성 경로의 핵심 요소인 rfbA, cpsC, cpsD, cps2G 유전자는 glycosyltransferase 및 polysaccharide polymerase를 암호화하여 숙주의 외부 스트레스 내성과 환경 적응성을 향상시킬 가능성이 있다[28,30,38]. 이와 함께 Region 2에서 확인된 신호전달 인자인 tyrosine phosphatase(ywqE_1)는 단백질 탈인산화를 통해 탄수화물 대사와 세포벽 합성에 관여하는 효소로 보고되었으며[31], 이러한 인자가 prophage 영역 내에 존재한다는 점은 cryptic prophage가 숙주의 대사 네트워크 조절에 영향을 미칠 수 있는 유전적 인자를 보유하고 있음을 시사한다.
결과적으로 L. plantarum WCFS1 유전체 내 prophage는 단순한 비활성화 잔존체가 아닌, 특정 조건에서 숙주의 유전체 안정성 유지 및 외부 스트레스로부터 숙주의 적응에 기여하는 유전적 모듈로서 기능할 것이라 판단된다. 이러한 결과는 향후 LAB 스타터 균주 선별 및 안전성 평가 과정에서 prophage 및 cryptic prophage의 존재 여부를 중요한 고려 요소로 포함해야 함을 시사한다.
