서 론
건강한 성인의 분변 내에는 약 400여 종 이상의 다양한 세균이 공생하고 있다. 이러한 장내 세균들은 분변의 고형물 무게 중 40%–45%을 차지하며, 평균적으로 분변 gram당 약 3.3×1012 CFU/g의 균수를 보인다[1,2]. Bifidobacterium 속(genus) 미생물들은 Bacteroides, Fusobacterium, 및 Eubacterium 속 등과 함께 인간의 장내 우점 미생물로 손꼽힌다. Bifidobacterium 속 미생물들은 유아기 때에 장내 균총의 90%까지 차지하다가 점차 감소하여 성인이 되어서는 전체 균총 중 10%–20% 정로도 낮아진다[3].
Bifidobacterium 속의 세균들은 bifidobacteria로 불려지기도 하는데, 비피더스균은 오래전에 bifidus가 종(species)명으로 사용되었을 때 생겨난 bifidobacteria의 일본식 표현이다. 1900년 Tissier의 학위논문에는 유아 분변에서 Y 형태의 특이한 모양의 세균을 관찰하였으며, ‘갈라진 형태(bifurcated forms)’의 미생물을 Bacillus bifidus로 명명하였다. 또한, Tssier는 태어난 지 3일이 지나면 이 미생물이 관찰되었으며, 모유를 먹은 유아에서 주로 존재하는 장내 미생물이라고 보고하였다[4–6].
Bifidobacteria는 편성혐기성균으로 일반적으로 37℃–41℃의 온도에서 최적의 생육 환경을 가진다[7]. 그리고 pH 6.5–7.0에서 왕성하게 성장하며 fructooligosaccharide, glucose, maltose, raffinose 등이 존재하는 환경에서 비교적 잘 자란다. 최근에는 bifidobacteria가 알레르기 및 감염성 설사 증상 완화와 같은 효과가 있음을 임상적으로 입증되었다. 오래전부터 여러 생물 자원으로부터 bifidobacteria를 분리하고자 하는 노력은 지속되어 왔는데, 이들 균주만을 분리하기 위해서 유기산, 항생물질, 올리고당, 및 박테리아파지 등을 사용한 다양한 선택배지가 연구되었다[8].
Bifidobacteria 균주 선발을 위한 최초의 선택 배지는 1951년 Frisell이 NaN3를 첨가한 Tomato-agar Orla-Jensen medium에서 기원을 찾을 수 있다. 그 후 1957년 Haenel과 Mueller-Beuthow가 sorbic acid을 첨가한 Sorbic acid-culture medium에서 bifidobacteria를 분리하였다. 1960년 Ochi는 항생제인 neomycin을 첨가한 EG medium을 사용하여 bifidobacteria를 선별적으로 분리하였으며, 1961년 Mitsuoka 연구팀은 neomycin, paromomycin, LiCl, Na(C2H5COO)를 첨가한 BS medium Ⅰ과 Ⅱ를 사용하여 분변에서 bifidobacteria를 분리하는데 성공하였다. 그 후 Finegold 연구 그룹은 1971년 kanamycin을 함유한 Standard blood agar medium을 이용하여 bifidobacteria 생균수를 평가하였으며, Teraguchi et al.(1978)은 항생제 3종(neomycin, paromomycin 및 nalidixic acid)과 lithium chloride를 BL(blood-liver) 배지에 혼합한 bifidobacteria 선택배지(BL-NPNL)를 제조하였는데, 이 배지는 bifidobacteria가 함유된 발효유제품에서 다른 유산균의 성장을 억제시키고 bifidobacteria의 생균수를 평가하는데 효과적인 선택배지로 현재까지 많은 연구에 사용되고 있다[9].
국내에서도 분변과 유제품에서 bifidobacteria를 분리하고자 하는 다양한 연구가 시도되었다. Ji et al.은 galacto-oligosaccharide와 propionic acid를 selective agent로 첨가하여 분변에서 순수한 bifidobacteria를 분별할 수 있었다고 보고하였는데, 이 배지는 식품 중에 함유되어 있는 bifidobacteria의 선택배지로 식품공전에 제시되어 있다[10]. Shin et al.은 bifidobacteria를 함유한 발효유제품에서 유산균 특이적인 bacteriophage를 사용하여 bifidobacteria만을 효율적으로 검출하는 방법을 최초로 보고하였다[11]. 또한, Rada와 Koc은 Methicillin-resistant Staphylococcus aureus에 효과적인 항생제인 mupirocin을 혐기성미생물의 생육배지인 Wilkins-Chalgren broth에 첨가하여 발효유제품에서 bifidobacteria를 선택적으로 분리할 수 있었다고 보고하였다[12]. 현재까지도 다양한 종류의 미생물 생육억제 물질을 단독 또는 혼합한 bifidobacteria 선택배지에 대한 연구는 계속되고 있다[13].
이에 본 연구는 bifidobacteria 연구에서 많이 사용되는 BL-NPNL 배지[14]와 항생제를 배제하고 L-cystein을 첨가한 MRS-C(pH 5.0) 배지, 그리고 L-cystein과 mucin을 첨가한 BHI-CM 배지를 각각 사용하여 성인 분변 시료로부터 bifidobacteria의 분리 특성을 비교함으로써 bifidobacteria를 위한 검출 배지 개발에 기초자료를 제공하고자 수행하였다.
재료 및 방법
25세의 건강한 성인 여성의 분변 시료를 유동 파라핀이 중층된 blood-liver(BL) broth를 사용하여 혐기적 상태로 유지시킨 후 bifidobacteria 분리 시료로 사용하였다. Bifidobacteria 분리용 배지로는 BL-NPNL 배지[14], 0.05% L-cysteine을 첨가한 후 pH를 5.0으로 조정한 MRS-C 배지, 그리고 0.05%의 L-cysteine과 0.5% mucin을 첨가한 BHI-CM 배지를 제조하여 사용하였다. 이후 분변 시료를 혐기성 희석액으로 희석하여 각 배지에 접종한 다음 steel wool법으로 혐기성 배양기(FA-6R, Hirayama Manufacturing, Saitama, Japan)에서 37℃, 72시간 동안 배양하여 유백색의 집락을 선발하였으며, BHI-CM 배지에서는 mucin을 분해하는 집락을 선발하였다.
선별한 균주들을 동정하고자 16S rRNA gene sequencing을 수행하였으며, 균주들의 유전자는 the universal rRNA gene primers을 통하여 증폭하였다. 그리고 증폭된 유전자를 Solgent (Daejeon, Korea)에 위탁 분석하였고, NCBI blastsearch(https://www.ncbi.nlm.nih.gov)를 이용해 sequence 결과를 확인하였다.
결과 및 고찰
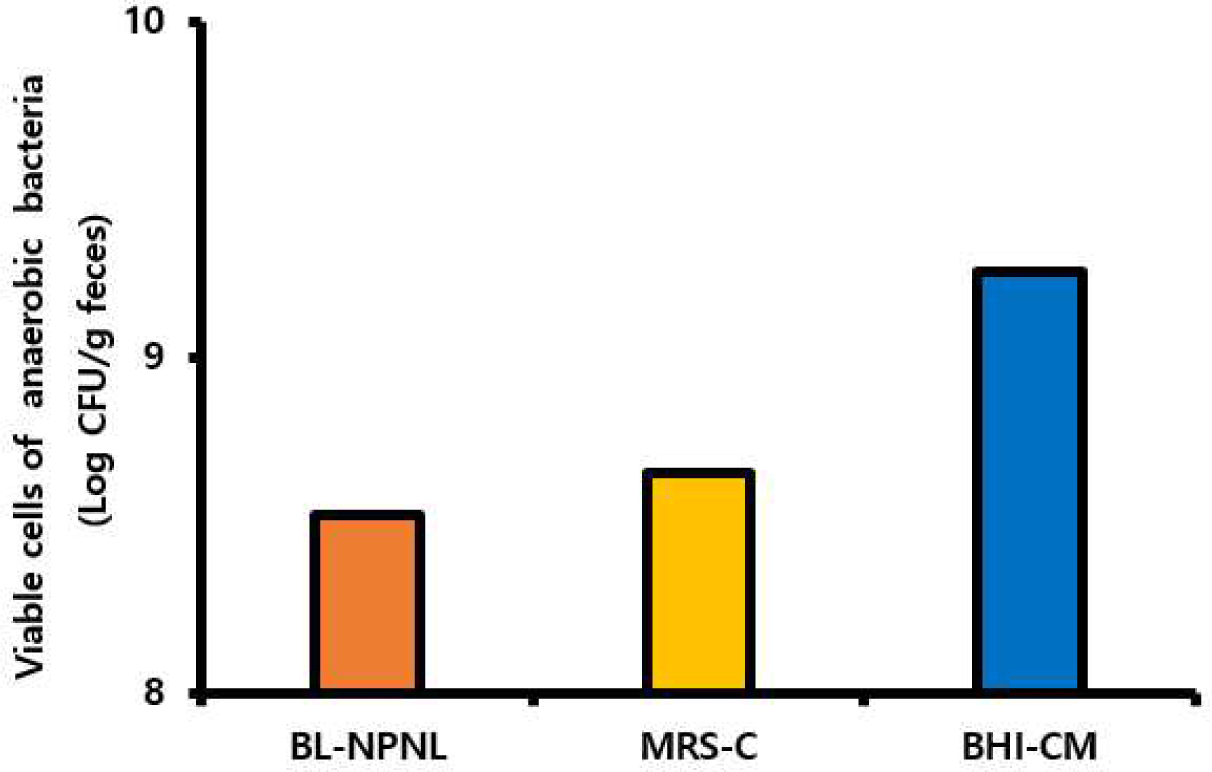
Fig. 1은 분변시료를 BL-NPNL, MRS-C, 및 BHI-CM 배지에 각각 도말하여 혐기조건에서 평가한 총생균수를 나타낸 것으로 각각 8.5, 9, 및 9.25 Log CFU/g feces로 나타났다. 특히, BHI-CM 배지에서 가장 높은 생균수를 보였는데, 이는 mucin 분해 혐기성 세균들을 포함하여 Gram 양성 및 음성 세균들도 같이 검출되었기 때문이다. 이때 Bifidobacterium 속 균주를 포함한 장내 공생세균의 게놈에는 다른 환경에서 서식하는 미생물의 게놈에 비하여 mucin 분해에 관여하는 유전자를 많이 발견할 수 있는데, engBF(endo-α-N-acetylgalactosaminidase)와 afcA(1,2-α-l-fucosidase) 유전자가 바로 그 예이다. 이러한 유전자들은 장내 공생 세균들로 하여금 열악한 장내의 환경에서 생존하기 유리하도록 적응 가능하게 만들어주었다[15].
각 배지에서 유백색 집락 약 200여개로부터 16S rRNA를 분석한 결과, 실험에 사용한 배지에서 확인된 집락은 총 31종의 서로 다른 species로 구별되었다(Table 1). BL-NPNL 배지에서는 B. longum과 Bacillus 속의 균주들이 다수 검출되었는데, 복합 항생제 첨가에 의하여 그 외 미생물은 생육이 억제된 것으로 추정되었다. MRS-C 배지에서는 B. longum과 B. animalis 균주들을 분리할 수 있었으며, 이외에도 Lactobacillus acidophilus와 Enterococcus avium 등의 유산균도 함께 검출되었다. BHI-CM 배지의 경우, 분변 시료에서 B. longum, B. faecale 및 B. animalis 3종의 bifidobacteria를 검출할 수 있었다. 흥미롭게도 Gram 양성 균주들 위주로 검출되었던 BL-NPNL 배지와 MRS-C 배지와는 다르게, BHI-CM 배지에서는 Parabacteroides 속과 Bacteroides 속 등의 Gram 음성 절대 혐기성 세균들이 다수 검출되었다. Nam et al.은 한국인 20명의 분변 메타게놈을 분석한 결과 개인별 차이가 많이 있었으며, 주로 Bacteriodes 속, Clostridium 속, Eubacterium 속, Faecalibacterium 속, Ruminococcus 속 등 다양한 미생물을 확인할 수 있었다고 보고하였다[16].
본 연구에서는 동정된 균주들 중에서 Bifidobacterium 속 균주 4종인 B. longum subsp. suillum, B. longum, B. animalis subsp. lactis, 및 B. faecale를 SY517, SY526, SY615 및 SY827로 각각 명명하였으며, 추가적인 배양 시험을 진행하였다.
최종 선발된 SY517, SY526, SY615, 및 SY827에 대한 균주 특성은 Table 2에 제시된 바와 같으며 Gram 양성, Catalase 음성 및 KOH test 음성으로 확인되었다[17].
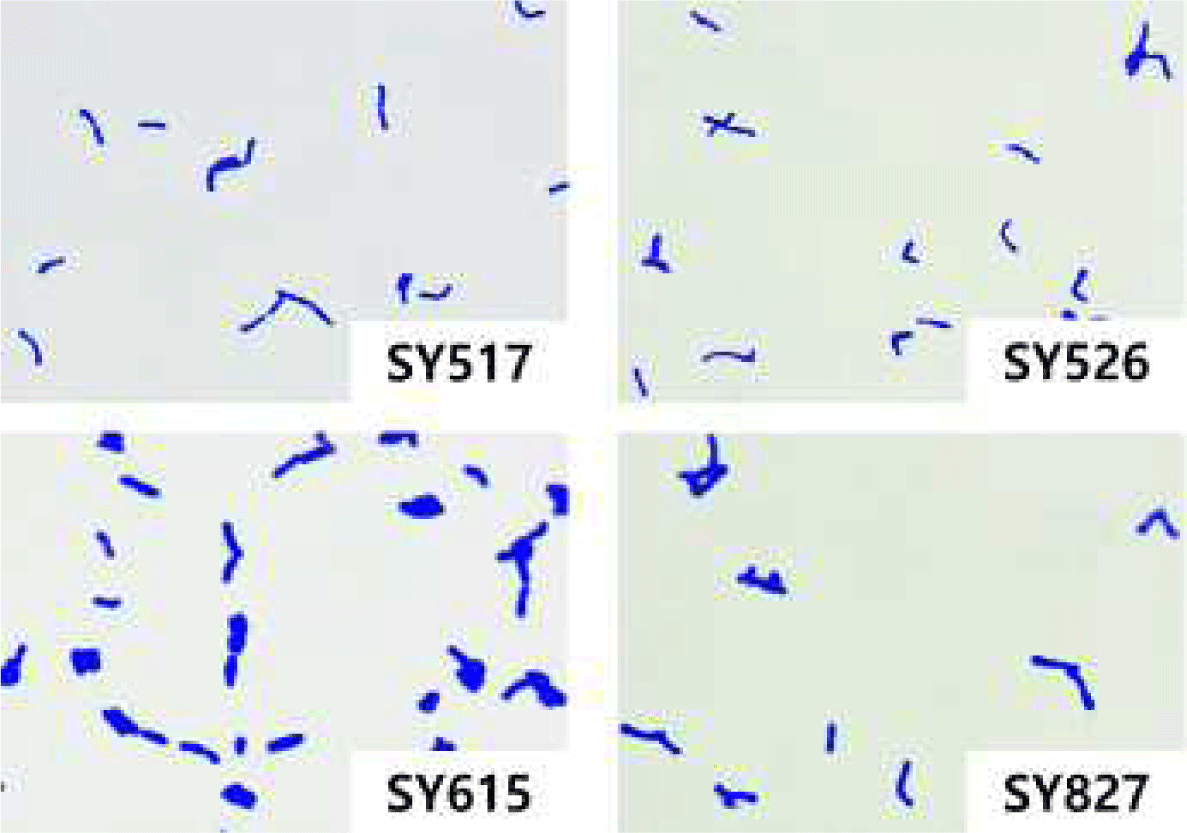
선발 균주들은 광학 현미경으로 세포모습을 확인한 결과, 모두 막대, V, Y, 곤봉 등의 bifidobacteria 특징적 성상을 보였다(Fig. 2). 특히, SY517은 간상 형태와 V자 형태가 많았고, SY 526은 V자와 곤봉형태가 다수 관찰되었다. 그리고 SY615는 짧은 rod형이 많았으며, SY827은 cocco-bacilli와 곤봉형의 모양이 관찰되었다.
실험에 사용된 3종의 배지에서 선발된 4종의 bifidobacteria를 배양한 결과, BL-NPNL 배지에서는 SY517과 SY526만이 집락을 형성하였는데, SY517은 작고 유백색의 둥근 집락 형태를 나타냈으며 SY526은 크기가 크고 노란색 원반 형태의 집락 모습을 보였다. 그리고 MRS-C 배지의 경우, SY526과 SY615가 검출되었는데, SY526의 경우 둥글고 작은 투명한 집락을 형성하였으며 SY615는 작고 둥근 백색의 집락으로 자라났다. BHI-CM 배지에서는 모든 Bifidobacterium 속이 검출되었는데, SY517의 경우 작은 백색 집락을 형성하였고 SY526은 노란색 집락 형태를 보였다. 그리고 SY615는 아주 작은 집락으로 관찰되었으며, SY827은 유백색이 아닌 표면이 매끄럽고 투명한 집락의 형태를 보였다(Fig. 3).

Table 3에 제시한 Bifidobacterium species의 genome은 현재까지 보고된 유전체 정보를 바탕으로 요약 정리한 것으로 species에 따라 1.7 Mbp에서 3.3 Mbp까지 다양한 분포를 나타내었다. Bifidobacteria의 유전적 다양성은 서식하는 환경 조건에 따라 차이를 보이는 것으로, 곤충의 장내에서 분리한 B. indicum은 식물에 존재하는 탄수화물의 대사가 가능한 다양한 enzyme을 보유하고 있는 것으로 알려져 있다. Bifidobacterium 종들은 서로 다른 숙주의 공생 박테리아로부터 수평적으로 유전자를 전달받아 상이한 유전적 방향성을 가지고 환경 특이적으로 진화를 거듭해 왔다[18]. 유아의 분변에서 분리한 B. longum subsp. infantis는 모유 올리고당을 대사할 수 있는 α-fucosidase, α-sialidase, β-galactosidase 및 β-N-hexosaminidase의 유전자를 가지고 있으며, 성인의 분변에서 유래된 B. adolescentis와 B. longum subsp. longum은 효과적으로 식이 탄수화물을 대사할 수 있는 다수의 유전자를 가지고 있다[19]. 환경적 차이에 의한 Bifidobacterium 속 균주들의 다양성은 진화 적응을 해왔음을 의미하는 것으로 향후 전장유전체 분석과 대사체 분석을 통하여 환경 특이성과 유전체와의 연관성이 규명될 것이다.
4종의 선발 bifidobacteria에 대한 배지별 생균수를 평가한 결과는 Fig. 4에 나타난 바와 같다. SY517과 SY526는 BL-NPNL 배지에서 배양 36시간에 OD600값으로 0.61과 0.8 정도까지 성장하였고 이때 배지의 pH는 3.8과 3.9까지 저하되었다. Lim et al.의 연구결과에 의하면 B. longum 균주의 neomycin과 paromomycin에 대한 최소억제농도(minimum inhibition concentration; MIC)는 각각 평균적으로 200 mg/mL–400 mg/mL와 400 mg/mL–800 mg/mL인 반면에, B. infantis와 B. animalis 균주들의 경우, neomycin과 paromomycin의 MIC는 둘다 100 mg/mL–200 mg/mL의 범위를 보인다고 보고 하였다. 따라서 BL-NPNL 배지에서 B. longum 균주들이 다른 종보다 생육억제를 받지 않은 이유는 neomycin과 paromomycin에 높은 MIC 때문으로 추정되었다[20]. 따라서 BL-NPNL 배지는 SY517과 SY526과 같은 B. longum 균주들을 선택적으로 분리하기에는 적절하지만 SY615와 SY827와 같은 균주는 분리가 어려울 것으로 판단되었다.
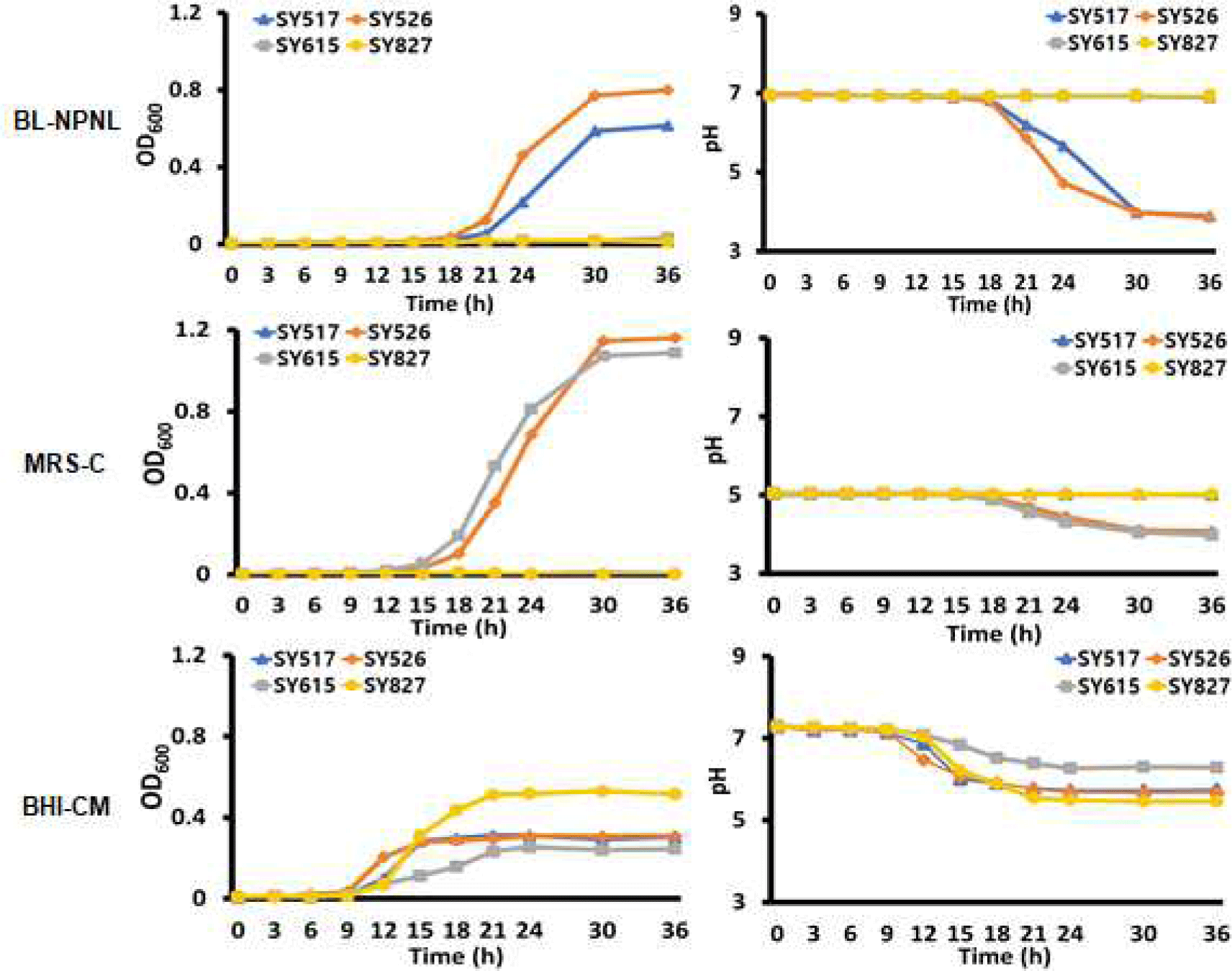
MRS-C 배지의 경우 SY517과 SY827 균주들은 배양 36시간까지 생장이 낮았으나, SY526과 SY615 균주들은 배양 36시간에 OD600가 각각 1.16과 1.098의 수준으로 높은 생장을 보였다. 이는 정상범위보다 낮은 pH에서는 세포막에 존재하는 H+-ATPase가 활성화되어 유산균이 낮은 pH에 적응을 할 수 있도록 하기 떄문으로 사료된다[21]. 특히 B. longum과 B. animalis는 일반적으로 내산성이 높은 미생물이기에 초기 pH를 5.0으로 조정한 MRS-C 배지에서 H+-ATPase가 활성화되어 빠른 생육이 가능했을 것이다[22].
SY827 균주는 BL-NPNL과 MRS-C 배지보다 BHI-CM 배지에서 가장 높은 생장을 보였다. BHI-CM 배지의 경우 배양 36시간에 SY827 균주의 OD600는 0.52까지 성장하였으나, SY517, SY526, 및 SY615 균주들은 각각 0.305, 0.308 및 0.245을 나타내었다. 이는 Bifidobacterium 속 균주들이 mucin을 에너지원으로 이용할 수 있도록 1,2-α-l-fucosidase, endo-α-N-acetylgalactosaminidase 등과 같은 효소를 분비하는데, SY827 균주가 다른 균주들보다 효소의 활성이 높아 이러한 생장을 보였다고 생각되는 까닭이며 추가적인 연구로 규명되어야 할 것이다[23].
이처럼 분변을 포함한 다양한 환경 시료로부터 bifidobacteria를 분리하고자 할 때에는 BL-NPN 배지와 MRS-C 배지보다 BHI-CM 배지를 사용하면 더욱 다양한 종류의 bifidobacteria 분리가 가능할 것으로 추정된다. 그러나 BHI-CM 배지는 선택배지가 아니기 때문에 시료에 함유되어 있는 Gram 음성 세균이나 다른 종류의 미생물이 자랄 수 있음을 유의해야 할 것이다.
Meta genome 분석은 장내 microbiome을 비롯하여 다양한 환경에서의 미생물군을 파악할 수 있는 장점이 있다. 그러나 유용한 균주를 산업적으로 이용하고자 할때에는 생균 상태로 확보하는 것이 선행되어야 한다. 본 실험결과에서 나타났듯이 Bifidobacterium속 균주들은 여러 배지에서 서로 상이한 생장을 보인다. 따라서, 실험자가 필요로 하는 균주를 선발하기 위해서는 각 species의 특성에 맞는 배지를 적절히 사용하는 것이 필요하다. 여기에 16S rRNA 분석 결과를 포함한 다양한 생화학적 특성을 파악함으로써 선발 균주의 기초자료 확보가 된다면 산업적 활용성을 보다 높일 수 있을 것이다.
