서 론
항생제는 사람, 동물, 식물의 세균 감염을 막기 위해 사용되는 중요한 방어 수단으로 의료, 축 농수산업 등을 포함하여 광범위하게 사용되고 있다[1]. 낙농산업에서도 항생제의 문제점이 예외가 될 수 없는데 특히 치료목적으로 사용되는 항생제가 우유에 잔류되었을 경우 사람이 섭취할 수 있다는 문제점뿐만 아니라 스타터를 이용한 유가공품의 숙성 및 발효에 영향을 미쳐 생산량 저하 등의 경제적 손실의 문제를 초래한다. 이러한 광범위한 항생제의 오남용은 슈퍼박테리아 등의 항생제 내성의 위기를 초래하였고, 그 결과 국내외적으로 새로운 항생제의 개발과 항생제 오남용 감소를 위한 노력에 관심이 높아지고 있는 가운데 항생제 내성균에 대한 우려는 높아지는 반면, 종종 간과되는 주요 타겟에는 항생제에 내성이 있는 persister 세포가 있다[2]. 세균이 항생제에 내성을 가지게 되면 항생제로부터 살아남을 수 있는데, perister는 세균 감염을 재발시키는 등 세균박멸에 어려움을 겪도록 한다. 따라서 persister의 형성과 재개의 매커니즘을 잘 이해한다면 항생제 내성률 감소 및 지속적인 세균 감염을 근절할 수 있는 새로운 접근법이 개발될 수 있을 것이다[3]. 퍼시스터 세포는 박테리아 세포가 스트레스에 노출되었을 경우 세포의 대부분이 휴면 상태가 되고, 이 휴면 세포는 스트레스 요인이 제거되면 다시 소생한다는 특징을 가진다. 병원성 세균의 경우 이러한 휴면상태를 통해 스트레스를 극복하고 감염성을 다시 획득할 수 있다는 점을 고려하였을 때, 항생제 내성률의 감소라는 목적을 위해서 이러한 휴면 상태의 세포를 박멸하는 방법을 찾아내는 것이 매우 중요하다. 이러한 목적을 가지고 지난 몇 년 동안 여러 연구 그룹을 통해 persister를 죽이는 많은 인돌 관련 화합물이 발견되어 왔다. 따라서 이 논문은 persister의 형성과 소생에 관한 최근 연구 결과들의 요약과 함께, 병원성 및 비병원성 persister를 죽이는 인돌과 치환 인돌의 기능을 설명하고자 한다.
본 론
Persister는 유전적 변화 없이[4] 대사를 정지시켜 발생하는 스트레스 내성 세포이다[5–8]. 처음으로 Persister가 알려진 것은 휴면 상태의 Staphylococcus aureus가 페니실린에 내성이 있음을 보여주는 연구 결과[5,6]로 규명되었다. 세포 집단의 소그룹에서 일어나는 persistence와 달리 resistance는 항생제가 존재할 때 세포가 성장하도록 하는 유전적 돌연변이가 발생하여 일어나고, tolerance는 세포가 느리게 성장하면서 이 전체 세포 집단이 항생제에 대한 감수성이 감소된다[9,10]. Persister 세포는 영양 제한, 항생제, 산성 및 산화적 스트레스에 의해 나타날 수 있다[11,12]. 거의 대부분의 세포들은 영양이 고갈되어 있기 때문에[13], persister 세포는 세균과 고세균류에서 발생하는 보편적인 휴면 상태이다[14]. Persistence가 자연적으로 발생하기도 하지만[15], 주로 환경의 스트레스에 반응하여 발생한다[7,16–22].
ppGpp는 persister와 관련이 있는 인자로 알려져 있으며[23–26], 신호인자로서 persister를 형성하는 역할을 한다는 것은 이미 많은 연구를 통해 알려져 있다[23–26]. 하지만 최근까지도 ppGpp의 persister 형성 기작은 명확히 규명되지 않고 있다.
ppGpp와 persister 사이의 관계를 명확히 하기 위해서는 ppGpp의 박테리아 세포 대사 조절 기작을 알아야 한다. 먼저 세포는 스트레스 상태를 견디기 위해 구아노신 4인산 또는 구아노신 5인산(ppGpp)을 합성하여 복제, 전사, 번역을 감소시킨다[27]. 또한 ppGpp는 DNA primase를 억제하여 DNA 복제를 느리게 만들고[27], RpoS(sigmaS, 정지기의 스트레스 반응 시그마 인자)와 RpoE(sigmaE, 세포질에서 잘못 접힌 단백질에 대한 스트레스 반응 시그마 인자)를 자극하여 전사를 느리게 한다[28]. 또한 퓨린 뉴클레오타이드의 합성을 방해하고[29] 핵산 분해효소 PpnN을 활성화하여[30] 퓨린 항상성을 조절한다. 또한 ppGpp는 리보솜의 생산을 줄여 번역을 느리게 만들기도 한다[31].
특정 단백질의 활성도 ppGpp에 의해 직접적으로 감소된다. 예를 들어, ppGpp는 GTP 가수분해 효소(GTPase)에 결합하여 GTPase의 활성을 방해한다[27]. ppGpp는 비활성화 상태의 100S 리보솜을 활성화하는 GTP 분해 효소인 HflX 단백질[32]에 결합하여 비활성 리보솜의 활성화를 억제한다[32,33](Fig. 1A). 또한 ppGpp는 30S 리보솜 소단위의 생성에 관여하는 GTPase Era(필수 GTPase)를 억제한다[34].
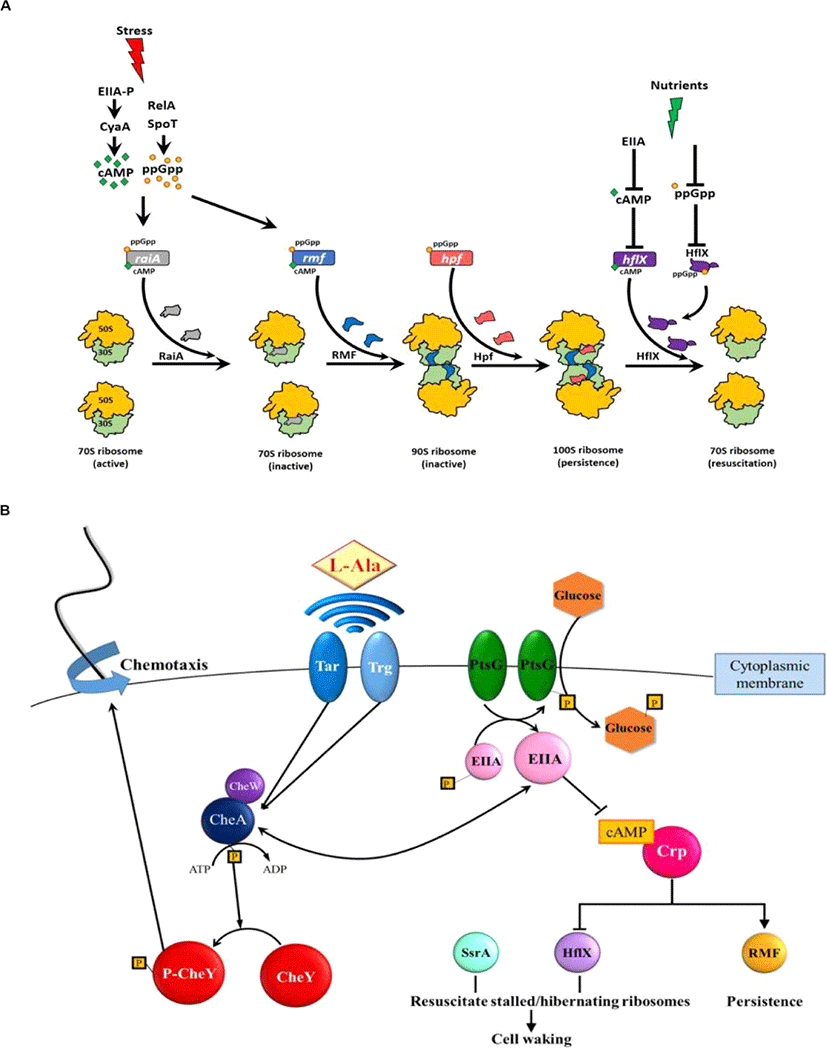
결정적으로 persister를 형성하기 위해 ppGpp는 (i) 70S 리보솜을 비활성화하는 리보솜 조절 인자(RMF)를 암호화하는 rmf, (ii)정지 유도 인자(Hpf)를 암호화하는 hpf, (iii)리보솜과 관련된 방해인자(RaiA)를 암호화하는 raiA를 유도하여 리보솜을 비활성화한다.
다른 많은 연구에서 ppGpp가 Toxin/Antitoxin(TA) 시스템의 Toxin을 활성화하여 persistence를 형성하는 메카니즘을 규명하려 시도했으나 여전히 규명되지 못하고 있다[35–37]. 반면 박테리아가 스트레스에 노출되면 ppGpp가 유도되고 70S 리보솜을 비활성 100S 리보솜으로 전환하여 Persister를 형성하는 리보솜 이량체 Persister 모델(PRDP, Fig. 1A)이 제안되었다[14,22]. 이를 뒷받침하는 실험결과로 (i) persister의 리보솜은 대부분 비활성 100S 리보솜이고, (ii) RMF, Hpf, RaiA의 비활성은 persister의 형성을 감소, persister의 소생을 증가시켰으며 (iii) 단일세포 분석을 통해 persister의 소생이 ppGpp의 농도에 영향을 받지 않는다는 것을 발견했다[14]. Persister 형성에서 TA 시스템과 persistence 사이의 관계가 충분히 증명되지 않았기 때문에 이PRDP 모델은 TA 시스템에 의존하지 않는다[8,38,39].
Persister 세포는 ppGpp의 농도와 관계없이 형성되므로[25] PRDP 모델에서는 ppGpp 없이 RMF와 Hpf를 활성화하는 데 cAMP의 역할이 포함되어 비활성 100S 리보솜이 형성된다(Fig. 1A). 특히 영양 부족(포도당 고갈)은 cAMP를 증가시키고 rmf[40]와 raiA[41]를 유도한다. 또한 cAMP는 hflX의 활성을 억제한다[42]. 따라서 cAMP는 persister 형성에서 ppGpp와 비슷한 역할을 하는데, 이 두 세포 신호(ppGpp와 cAMP)의 증가가 리보솜 비활성화와 세포 소집단의 persistence 형성으로 이어진다.
persister의 소생에서(Fig. 1B), 단일세포분석방법을 통하여 그들이 외부 영양소를 인식하는 다양한 방법으로 소생한다는 것을 처음으로 입증하였다. persister가 깨어나는 비율은 활성 리보솜의 수에 의존한다. 이후 persister 소생의 다양한 특성은 많은 연구에 의해 검증되었다[43,44]. Single cell 분석과 전반적인 E. coli 단백질에 대한 연구를 통해, persister의 소생은 외부의 영양소를 인식하며 시작되며, persistence의 소생에 특화된 단백질은 딱히 존재하지 않는다(Fig. 1B)[45]. 2차 메신저 cAMP의 농도가 감소되면서 외부 영양소의 존재(즉, 신호)가 세포질로 전파되는데, 이때 cAMP의 농도 감소는 mRNA 상에 정지된 100S 리보솜을 활성화시키고 이때, HflX이 비활성 100S 리보솜을 활성화하도록 한다[45]. 소생된 세포는 인식된 영양소를 향한 주화성 운동(chemotaxis)을 시작하는데, 이는 애초에 영양소의 고갈이 persistence를 유발했기 때문이다[45]. 따라서 Wood 그룹에서는 소생에서 특정한 신호와 이 신호가 어떻게 세포 외부에서 감지되는지, 외부의 신호가 어떻게 2차 메신저를 통해 세포 내부로 전달되는지, 그리고 세포가 persister에서 깨어나서 영양소에 반응하여 어떠한 화학 작용을 시작하는지를 규명하였다[45].
PRDP 모델(Fig. 1A)은 persister의 형성이 스트레스에 대한 세포의 조절 반응(elegantly regulated response)이라는 것을 시사한다. 이에 대한 실험적 근거로, 자연적인 persister는 많지 않지만[15] 다양한 형태의 환경적 스트레스(항생제, 과산화수소, 산 등)가 지수기의 세포 집단 거의 대부분을 persister로 전환시킨다[7,11]. 비슷하게 PRDP 모델은 persister의 소생 또한 자연적인 현상이기보다 고등의 환경 반응이라는 것을 시사하고, 아미노산 알라닌에 대한 persister 소생 연구결과가 이를 증명한다[45]. 거의 모든 세포가 영양 고갈을 겪고 그 스트레스 환경을 견디기 위해서 휴면 상태가 필수적이기 때문에 세포가 persister 형성과 소생에 고등의 조절을 요구하는 것은 합리적인 현상이다. 결정적으로, PRDP 모델은 persister에서 “표현형의 전환”이 비활성화된 리보솜 수에 의해 결정된다는 것을 시사한다. 따라서 스트레스 조건에서 비활성 리보솜을 일정 기준만큼 가진 일부 세포만이 persister가 된다[21,22]. 즉 모든 세포가 비활성 리보솜의 충분한 비율을 갖지는 않으므로 정지기의 세포가 모두 persister인 것이 아닌 것이다.
RMF가 (i) ampicillin[14], ciprofloxacin[14], netilmicin[46], gentamicin[47], 산[48], 삼투 스트레스[49] 및 영양 고갈[50,51] 등 수많은 스트레스 환경에서 E. coli의 persistence를 극도로 증가시키는 것이 관찰되었기 때문에, PRDP 모델은 다양한 스트레스 환경으로부터 persister가 형성되는 방법에 일반적으로 적용이 가능하다. RMF[41]와 HflX[52]는 거의 대부분의 세균 내에서 잘 보존되어 있고, Hpf는 여러 생물계(원핵생물 및 식물)에 널리 퍼져 있으므로[53], PRDP 모델은 아마 E. coli 뿐만이 아니라 많은 종의 persister의 형성에 적용할 수 있을 것이다. 예를 들어 기회 감염성 세균 Pseudomonas aeruginosa도 persister를 형성하기 위해 ppGpp를 요구하고[24], 영양이 제한되었을 때 리보솜을 보호하거나 P. aeruginosa 또한 장기간 생존하게 하기 위해 Hpf와 ppGpp (RMF 제외)가 필수적이다[54]. 또한 ppGpp는 P. aeruginosa에서 hpf 발현을 유도한다[53]. Rhodospirillum centenum 낭포의 경우 세포가 활성화되기 위해 첫 번째로 암호화된 유전자들은 리보솜과 번역 시스템(개시, 신장, 종결)과 관련되어 있다[55]. 따라서 PRDP 모델은 많은 종의 휴면 상태를 규명 가능하다.
트립토판의 대사 산물인 인돌은 종내, 종간, 생물계 간 신호라는 점에서 다중 계층 신호(interkingdom signal)이다. 종내 신호로써 인돌은 특히 주로 저온에서[56] E. coli의 쿼럼 센싱을 조절한다[57]. 종간 신호로써 인돌은 병원성 인자 pyocyanin, rhamnolipid, 2-heptyl-3- hydroxy-4(1H)-quinolone, pyoverdine[58]을 없애고, 이를 합성하지 못하게 된 P. aeruginosa의 병원성을 감소시킨다. 이는 P. aeruginosa와 공생하는 E. coli의 생존 경쟁 능력 향상으로 이어진다[59]. 또 종간 신호로써, 인돌은 EHEC(장출혈성 대장균)를 공격하거나(음성 화학 작용) 생물막의 형성을 억제하고, EHEC의 이동성과 HeLa 세포에 대한 결합력을 감소시켜 그 병원성을 낮춘다[60]. 따라서 여러 연구들은 인돌이 항독성 물질로써 사용될 수 있을 것이라고 주장해왔고[58,61], 실제로 인돌은 기니피그의 폐결핵을 줄이고 폐 간극을 증가시킴으로써 P. aeruginosa의 병원성을 낮추는 데 성공적으로 이용되었다[58]. 12년 후, Sperandino의 팀은 인돌이 위장관에서 EHEC의 병원성을 줄인다는 것을 확인했다[62]. 인돌은 S. aureus의 병원성 또한 감소시켰다[63].
놀랍게도 인돌은 생물계 간 신호이기도 하다. 위장관 내 미생물에서 생성된 인돌은 사람의 상피 세포 접합 부위를 꽉 조여 병원균의 침투를 막는다[64,65]. 또한 우리는 인돌이 훨씬 강한 신호가 되기 위해 위장관에서 산소화 효소에 의해 수산화 될 것이라는 가설을 세웠다. 예를 들어 수산화된 7-hydroxyindole은 기존의 인돌보다 P. aeruginosa의 병원성을 더 효과적으로 없앤다[57]. 게다가 많은 인간과 식물의 호르몬이 인돌에서 파생되었기 때문에(indole-3-acetic acid, serotonin, melatonin, epinephrine), 인돌은 세포 호르몬의 전구체가 될 수 있다[66]. 생물계 간의 신호로써의 인돌의 역할을 보여주는 증거로 일부 식물(옥수수)이 파밤나방의 애벌레와 같은 곤충에게 경고하기 위해 인돌을 방출한다는 것이 있다[67,68].
그리고 인돌은 E. coli의 생물막 형성을 감소시키고[57,66,69–71], 생물막 내에서의 E. coli 생산량을 감소시킨다[69]. 또한 인돌이 TA 시스템 YafQ/DinJ[20]와 인산분해효소 DosP[19]를 조절하여 E. coli persister의 형성을 감소시킨다는 사실 또한 밝혀졌다.
Persister를 죽이기 위해 사용되는 화학물질을 (i) persister 세포의 형성을 막거나, (ii)persister 세포를 죽이거나, (iii) persister 세포를 소생시켜 항생제 감수성을 다시 획득하여 전형적인 항생제로 죽이는 세 가지 범주로 나눌 수 있다[72]. 이중에서 indigoid는 주로 막을 손상시켜 휴면 상태의 세포를 죽임으로써 persistence를 억제한다.
E. coli에 2mM의 인돌이 persistence를 감소시키고 이것이 ampicillin의 52배 효과라는 연구결과가 발표되었다[19,20]. 인돌이 persistence를 감소시킨다는 확실한 증거로써 동일한 인돌이 E. coli의 내열성 또한 감소시킨다는 흥미로운 결과와 함께 최근 발표되었다[73]. 광범위한 persister를 죽이는 인돌의 능력은 고세균 계통 Haloferax volcanii를 죽이는 능력(188배에 달하는 살균능력)에서도 나타난다[74].
Persister를 박멸하는 물질의 스크리닝을 통해서 halogenated-, methoxy-, methyl-, nitro-인돌을 포함한 36개의 인돌 파생 물질의 persister 살균능력이 측정되었다. 이 검사에서, 4-fluorindole, 7-chloroindole, 7-bromoindole, 5-iodoindole과 같은 할로젠 인돌이 E. coli persister를 죽이는 것이 발견되었다. 특히 5-iodoindole은 기존 E. coli 인돌보다 1,500배 강한 가장 효과적인 indigoid이다[75]. 5-iodindole은 S. aureus의 persister도 죽였지만 P. aeruginosa에게는 효과가 없었다[75].
최근 P. aeruginosa의 persister를 효과적으로 죽이는 치환 인돌 5-nitro-3-phenyl-1Hindol- 2-yl-methylamine hydrochloride(NPIMA)[76]이 발견되었다. NPIMA는 rifampicin을 먼저 처리하여 전사를 멈추고 지수기의 E. coli를 persister로 전환한 후[7,77], 최초로 persister에 1만여개의 물질을 직접 처리하는 스크리닝을 통하여 발견되었다[76]. NPIMA가 5-iodoindole[75]과 cisplatin[78]보다 E. coli의 persister를 죽이는 데 훨씬 효과적이라는 것이 밝혀졌다. 중요한 점은 NPIMA가 P. aeruginosa와 S. aureus의 persister도 죽인다는 점이다. 결정적으로 NPIMA가 persister를 죽이는 작용이 세포막 손상으로 인한 것이라는 것이 밝혀졌다[75]. 나아가 NPIMA에 대한 E. coli의 내성은 일주일동안 거의 발생하지 않았으며, P. aeruginosa와 S. aureus의 감염 모델에도 NPIMA가 효과적이라는 것이 밝혀졌다[75].
더 나아가, 인돌 유래 물질은 항생제나 금속 물질에 결합해 persister의 억제능력을 증가시킨다. 예를 들어, tobramycin과 결합한 5-methylindole은 methicillin에 내성이 있는 S. aureus와 Staphylococcus epidermidis의 persister를 죽인다[79]. 5-nitroindole은 E. coli, P. aeruginosa, 그리고 Entrobacter tabaci의 persister를 죽이며, 구리 또는 아연 나노 입자와 결합했을 때 그 효과가 더욱 증가한다[80].
결핵으로 150만 명의 사람이 매년 사망하는 만큼[81], mycobacteria의 persister 박멸 가능 물질의 발견이 필수적이며 이에 치환 인돌 N-[(6-trifluoromethyl)-1H-indol-2-ylmethyl] cycloocctanamine(IMA6)이 Mycobacterium의 persister를 죽이는 것이 확인되었다[82].
Persister를 죽이는 것 이외에 인돌은 다른 특징이 있다. 인돌은 다른 세포가 휴먼상태에서 소생하는 것을 억제하고, 선택적으로 E. coli 세포를 휴면 상태에서 깨어날 수 있도록 한다[83]. 구체적으로, 인돌은 E. coli의 소생에는 아무런 영향도 미치지 않았지만 P. aeruginosa persister가 소생하는 것을 억제하였다[83]. 더 나아가 E. coli가 P. aeruginosa와의 경쟁에서 우월하게 만든다[83]. 생리학적 농도에서 인돌은 휴면 또는 비휴면 상태의 P. aeruginosa 세포에 독성이 없으므로[83], 소생 억제는 독성이나 형성된 P. aeruginosa의 persister 수 차이 때문이 아니다. 유감스럽게도 인돌이 persister 소생을 억제하는 기작은 아직 밝혀지지 않았다.
이러한 인돌의 특징은 E. coli에게 다른 경쟁자들보다 성장에 이점을 제공할 것이고, 이것이 E. coli에서 인돌이 0.7mM의 높은 수준으로 분비되는 주요 원인 중 하나일 수 있다[70]. 이 결과는 생리학적으로도 관련이 있는데, E. coli가 건강한 사람에게서 최대 12%[84], 중환자의 위장관에서 종종 발견되는 P. aeruginosa[85]와 함께 위장관에서 발견되기 때문이다. 인돌이 종 간 신호로써 P. aeruginosa의 쿼럼 센싱 관련 병원성 인자들을 감소시키기 때문에[58], 이러한 새로운 결과[83]는 E. coli에서 분비된 인돌이 P. aeruginosa의 병원성을 없애고 persister로부터의 소생을 막는다는 것을 의미한다.
결 론
E. coli로부터 분비된 인돌의 가장 중요한 측면은 쿼럼 센싱 신호로써 유전자 발현의 조절이 아니라 종 간 또는 생물계 간의 신호로써 주변에 영향을 미친다는 것이다. 인돌은 몇 가지 유전자만을 조절하는 반면[56,86], 패혈증을 막기 위해 상피 세포 접합부를 단단하게 만들거나[64], P. aeruginosa의 persister의 소생을 막고[83] EHEC[57,60]와 P. aeruginosa의 병원성을 감소시키는 등[58] E. coli와 공생하는 다른 균들을 제어하여 E. coli 숙주에게 유익한 환경을 만든다.
인돌이 persistence를 감소시킨다는 연구 결과를 이용하여[19,20], 많은 연구에서 persister를 죽이는 새로운 indigoid가 발견되고 있다. 따라서 우리는 항생제 내성의 심각성이라는 문제를 해결하기 위해 Persister 세포의 형성과 재개의 기작을 이해할 필요가 있으며, 이러한 과정에서 효과적으로 Persister를 제어할 수 있는 인돌유래물질 중 몇 가지를 효과적으로 사용할 수 있을 것이라 기대하며 이는 더 많은 연구가 필요할 것으로 보인다.
